一、项目简介
Gene expression-based genome-wide association study (eGWAS)是一种对公共数据库中相关的基因芯片及高通量测序实验数据的检索及整合,再通过统计方法计算其出现重复差异性表达的可能性,直接找到差异最显著的基因的方法。就其方法而言,其灵感来自于GWAS分析。常规的GWAS分析一般针对于SNP位点进行关联分析,而eGWAS是针对更为普遍的表达谱数据,以单个数据集为单位,进行类似SNP call的分析,然后对全部数据集利用类似meta-analysis的思路进行整合分析。eGWAS可以看作是针对芯片/高通量测序的meta-analysis。
二、实验设计方案
2.1 实验要求
2.2 差异基因获取
差异基因的获取采用如下的分析步骤:
从NCBI的GEO数据库中中搜集已发表论文的原始芯片及高通量测序数据
利用统计方法对每组数据单独分析,以鉴定每组数据的差异基因
计算di值,同时计算每个基因的p值。
筛选di ≥2 & p<0.01的基因作为候选的差异基因;
对于每个基因建立2x2列表,计算fisher’s exact test(fisher精确检验),统计p值。
按照fisher’s exact test p值大小对差异基因进行排序,并筛选最有可能的差异基因进行实验验证。
2.3 实验验证
Immunohistochemistry)等技术对筛选出的差异基因进行验证,以检验其准确性。具体实验可以参考相关的参考文献
三、数据分析结果
3.1 Chromosome plot散点图
以基因在染色体上的定位为横坐标, p值大小为纵坐标,绘制基因散点图示例。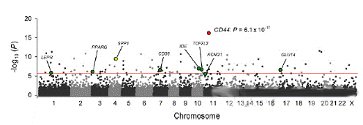
3.2 差异显著基因列表
与Chromosome plot散点图对应的,可以获得一个基因列表。该列表将根据p的大小进行排序。
3.3 差异显著基因功能分析
针对于3.2中的差异基因列表,进行GO(gene ontology)基因功能聚类分析。
图例如下所示: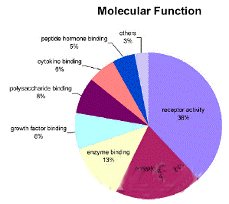
3.4 差异显著基因代谢通路分析
将筛选出的差异显著基因与相关的信号通路进行比较、整合,找出基因之间的相互关系,进行通路动态仿真,建立信号通路和生物功能网络。并根据统计检验方法(P-value)筛选显著显异的代谢通路
3.5基因网络分析
我们同时整合3种不同的相互作用关系建立基因网络:1)KEGG数据库中基因之间的蛋白互做、基因调控、蛋白修饰等关系;2)已有的高通量实验,如酵母双杂交等证实的蛋白-蛋白相互作用;3)已有文献报道的中提到的基因之间的相互作用。
四、参考文献
Keiichi Kodama,et al., Expression-based genome-wide association study links the receptor CD44 in adipose tissue with type 2 diabetes. PNAS, 109(18), 7049–7054