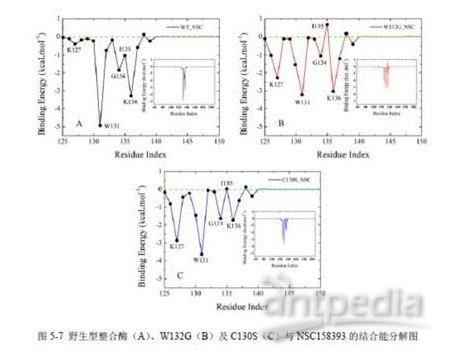
App Str-03 分子对接
随着X-射线衍射和多维NMR技术的发展,越来越多的蛋白质的三维结构被测定出来,但是蛋白质与其配体结合的复合物结构并不多。分子对接是分子模拟的重要方法之一,其目的是根据“几何匹配”和“能量匹配”原则研究两个或多个分子间的识别过程。利用分子模拟技术,能够对复合物结构进行较为准确的预测。分子对接是一种研究蛋白质受体与其配体相互作用与识别的重要手段,在药物设计、材料设计等领域有广泛的应用。比如可以用来从小分子数据库中搜寻并筛选出与受体生物大分子有较好亲和力的小分子,进行合成与活性测试,从中发现新的先导化合物。目前分子对接方法依然是基于受体结构的药物设计的首选方法。
分子对接思想的历史可以追溯到100年前Fisher提出的“锁钥模型”。根据“锁钥模型”,药物与体内的蛋白质即受体会发生类似钥匙与锁的识别关系,即一把“钥匙”开一把“锁”。这种识别关系主要依赖两者的几何匹配(图1),该模型又被称为“受体学说”。而随着时间的推移及生物学的发展,人们逐渐认识到药物与受体结合时,受体和配体的构象都会发生一定的变化以适应彼此的形状(图1)。1958年Koshland提出的分子识别过程中的“诱导契合”匹配概念[4],指出受体分子和配体分子在结合过程中,受体(或配体)分子将采取能与配体(或受体)分子最佳结合的构象,互相适应对方,从而达到最佳匹配。
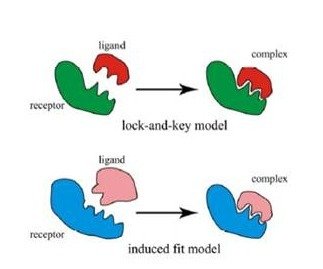
图1分子对接原理示意图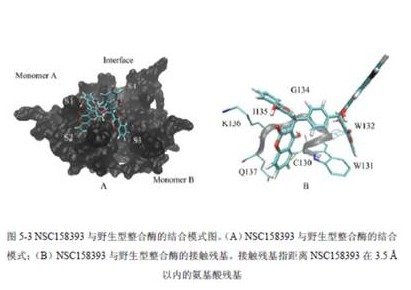
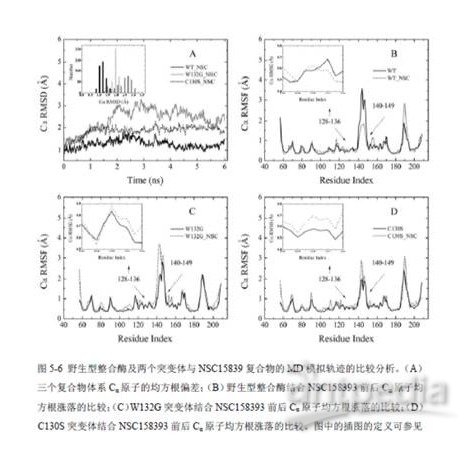
MD(分子动力学)模拟
七十年代末发展起来的MD模拟方法目前已经应用在许多研究领域,如生物分子的结构-功能关系、蛋白质结构预测、蛋白质折叠、酶反应机制、蛋白质-配体相互作用及动力学性质等[90, 91],并为合理药物分子设计等实际应用提供了坚实理论基础。
利用MD模拟,可对结合配体前后的蛋白质构象进行动态的分析,并给出相关的几何与物理参数。此外,MD模拟还是MM-PB/GBSA分析的基础。只有用MD模拟对体系进行长时间采样后,才能进行MM-PB/GBSA分析。
MM-PB/GBSA能量分析
MM-PB/GBSA是一种自由能计算方法。可将受体与配体之间的结合自由能分解到受体的每个与配体相接触的氨基酸残基上,从而确定在相互结合中起关键作用的氨基酸残基。