简化基因组甲基化测序MethylRAD-Seq
Methyl RAD技术(Wang et al., Open Biol., 2015)使用了甲基化修饰依赖性内切酶,如FspEI、MspJI、LpnPI、AspBHI等,此类内切酶会识别DNA上发生甲基化的胞嘧啶,在识别位置的下游隔一定距离切割双链。若DNA双链具有中心对称甲基化状态,则可以切割产生一个固定长度的双链DNA片段。对酶切产生的标签建库测序,即可进行甲基化位点的定性和相对定量分析。
欧易特色
文库构建简便快速,不经过片段大小选择,保证了实验的重复性
具有较强的灵活性,标签密度可控
标签长度一致,PCR扩增效率一致,测序深度均一
具有单碱基分辨率,可以做到甲基化位点的定性和相对定量分析
甲基化位点检测的假阳性率能够控制在0.50%以内(WGBS假阳性率在0.28%-0.50%)
提供标准和高级生物信息分析,可根据客户需求提供个性化数据分析
所需的样本DNA起始量较低
有参无参物种均可,既可用于未知甲基化位点的开发,也可用于不同样本间的甲基化位点的差异研究
推荐测序模式
● Hiseq X-Ten,PE150 ● 30 M reads/1 G基因组大小
案例展示
案例一 腭融合过程 EMT 机制调控研究
研究背景
腭突上皮细胞发生上皮-间质转化(EMT)是腭融合的关键环节,但是分子水平的调控机制,尤其是胚胎腭发育过程中的增强子甲基化的调控研究,仍不清楚。
研究内容
欧易生物携手汕头大学医学院第二附属医院,采用 Methyl RAD 技术对 6 个小鼠胚胎上鄂组织(实验组为全反式视黄酸处理;对照组为等量的玉米油处理)进行全基因组甲基化研究,通过筛选差异甲基化位点(CCGG 和 CCWGG)、GO 和 KEGG 富集分析,发现腭融合过程调控 EMT 的 3 个基因的增强子甲基化水平发生变化,可作为唇裂治疗的新表观遗传标记。

研究结果
1. 6 个小鼠胚胎上颚组织共获得 800M clean reads,其中 325M reads 具有酶切位点,234M reads 能比对到参考基因组。检出的差异甲基化位点 CCGG 和 CCWGG 位点分别为 17299 个和 2363 个(p < 0.05, log2FC >1),CCGG 位点平均深度不低于 29X,CCWGG 位点平均深度不低于 8X。
2. 聚类分析结果表明,差异甲基化位点在实验组的甲基化水平高于对照组的甲基化水平,另外,实验组和对照组的甲基化模式不同,存在明显的个体内和个体间甲基化差异。
3. 相比于 DNA 元件中的其他区域,大多数甲基化位点位于内含子和基因间区。
4. 筛选与腭部融合有关、并调控 EMT 的基因,并检测这些基因增强子的甲基化变化,结果 HDAC4、PDGFR 和 TCF7L2 的差异甲基化位点 CCWGG 位于内含子的非 CpG 岛。其中,HDAC4 增强子的差异甲基化位点发生了高甲基化,而 PDGFR 和 TCF7L2 增强子的差异甲基化位点发生了低甲基化,可作为腭融合治疗的靶标。
参考文献
Shu X, Shu S, Cheng H, et al. Genome-Wide DNA Methylation Analysis During Palatal Fusion Reveals the Potential Mechanism of Enhancer Methylation Regulating Epithelial Mesenchyme Transformation. DNA Cell Biol 2018; 37(6): 560-573. (IF: 2.236).
常见问题
1. Methyl RAD 技术中使用的是什么类型的酶?
以其中一种内切酶 FspEI 为例,其识别序列为 5’-CC-3’,且第二个胞嘧啶需发生甲基化,在此胞嘧啶所在链的右侧第 12 个碱基处、反向链右侧第 16 个碱基处进行切割,此时若反向链中心对称位置上存在同样的识别位点,则可切割产生 Methyl RAD标签,用于建库测序。
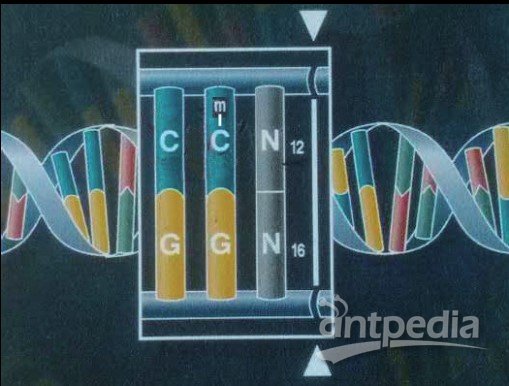
内切酶 FspEI 的识别及酶切位点
2. Methyl RAD检测到的甲基化位点数量如何?
Methyl RAD技术文章(Wang et al., Open Biol., 2015)中有Methyl RAD 和WGBS在拟南芥中检测到的CCGG和CCWGG两种甲基化位点的数量对比,从中可以看出二者的检出数量吻合度非常高。
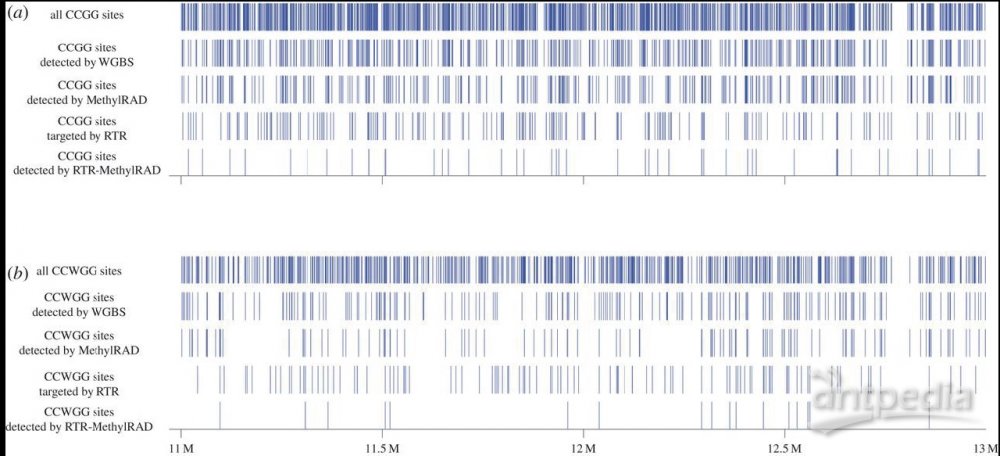
Methyl RAD、W GBS检测到的甲基化位点数量对比
(a、b图中的三行分别对应:基因组上预测出来的、WGBS检测到的、MethylRAD检测到的CCGG/CCWGG位点)
3. 如何调整标签密度?
Methyl RAD技术既可以增加标签密度,也可以降低标签密度。增大密度:可同时使用两种或两种以上的内切酶进行建库测序;降低密度:酶切后产生的片段为粘性末端,并且粘性末端的碱基是随机的。因此,在对酶切片段连接接头的过程中,可通过选择性接头特异地选择部分标签进行建库,以达到降低密度的目的。
参考文献
Wang S, Lv J, Zhang L, et al. MethylRAD: a simple and scalable method for genome-wide DNA methylation profiling using methylat ion-dependent restriction enzymes. Open Biol. 5, (2015). (IF: 4.822)