可以使用CRISPR–Cas编辑器在植物基因组中产生短插入和缺失,但是事实证明,很难在特定目标位点可靠产生较大缺失。
2020年6月29日,中国科学院遗传发育研究所高彩霞团队在Nature Biotechnology在线发表题为“Precise, predictable multi-nucleotide deletions in rice and wheat using APOBEC–Cas9”的研究论文,该研究报告了一系列APOBEC–Cas9融合诱导的缺失系统(AFIDs)的发展。 使用AFID-3在水稻和小麦原生质体(30.2%)和再生植物(34.8%)中产生的缺失大约是可预测的。该研究显示eAFID-3,其中AFID-3中的A3A被截短的APOBEC3B(A3Bctd)取代,产生更均匀的缺失。AFID可以用于研究调控区域和蛋白质结构域,以改善农作物。

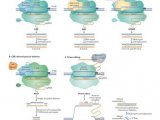
CRISPR–Cas9系统已广泛应用于基因组工程。简而言之,一个单一的指导(sg)RNA指导的Cas9核酸酶会产生染色体双链断裂(DSB),由于RuvC结构域的灵活切割,其末端可能同时变钝和交错。这些DSB主要通过非同源末端连接(NHEJ)进行修复,这导致频繁的短插入和缺失(indels)。但是,这些小插入缺失的异质性使其靶向小功能调节元件和结构域(例如顺式作用元件,microRNA等成为技术上的挑战。
有几种策略可用于生成目标较大的缺失,但都有缺点,例如,范围有限和所产生的缺失的不可预测性。特别是,成对的sgRNA策略需要两个适当间隔且有活性的sgRNA,这限制了它的范围,并经常在所需靶标之外产生长缺失。I-TevI:Cas9融合物切割目标DNA链并产生〜33至36 bp的缺失;因此,它们的长度相对恒定,但是很难生产,因为它们需要两个不同的识别位点,并且彼此之间有特定的距离。核酸外切酶(例如Trex2和T5)倾向于在Cas9介导的DSB的任一侧随机切除一个或多个核苷酸,从而导致一系列不可预测的缺失。
最近开发的prime编辑器使用工程化的Cas9切口酶-逆转录酶融合蛋白与工程化的prime编辑指导(peg)RNA配对产生所需的缺失,插入和核苷酸取代,但还需要提高其在植物中的效率。因此,需要新的策略来在整个基因组上产生可预测的多核苷酸缺失以进行精确编辑。
由许多胞嘧啶脱氨酶与Cas9切口酶(nCas9)和尿嘧啶糖基化酶抑制剂融合而成的胞嘧啶碱基编辑器已在许多生物中实现了C-to-T取代。当sgRNA与目标DNA链配对时,非目标DNA链会留为单链气泡,这有助于胞苷脱氨酶催化C到U的碱基取代。在碱基切除修复(BER)途径中,尿嘧啶DNA葡糖苷酶(UDG)识别U•G错配,切除基因组DNA中产生的尿嘧啶,并形成一个无碱基位点,导致嘌呤或无嘧啶位点裂合酶(AP裂合酶)使脱氨基链形成切口。)。
该研究报告了一系列APOBEC–Cas9融合诱导的缺失系统(AFIDs)的发展。 使用AFID-3在水稻和小麦原生质体(30.2%)和再生植物(34.8%)中产生的缺失大约是可预测的。该研究显示eAFID-3,其中AFID-3中的A3A被截短的APOBEC3B(A3Bctd)取代,产生更均匀的缺失。AFID可以用于研究调控区域和蛋白质结构域,以改善农作物。