我见过的相当一部分人,做质控时,一般也就就是跑个实验室或者公司的祖传代码,但对于软件所做的操作不求甚解,归根结底,是因为对测序流程中,接头是怎么加上去的不太了解。
现在我们接触的大多数二代测序数据,都是来自于illumina测序平台。这其中,大多数illumina文库的构建,是通过将接头连接到fragment DNA/cDNA的两端(但是Nextera方法除外,因为技术相对不常见,这里不深入展开)。 下图是一张很经典的加接头的示意图,图片下载于网页 http://tucf-genomics.tufts.edu/home/faq
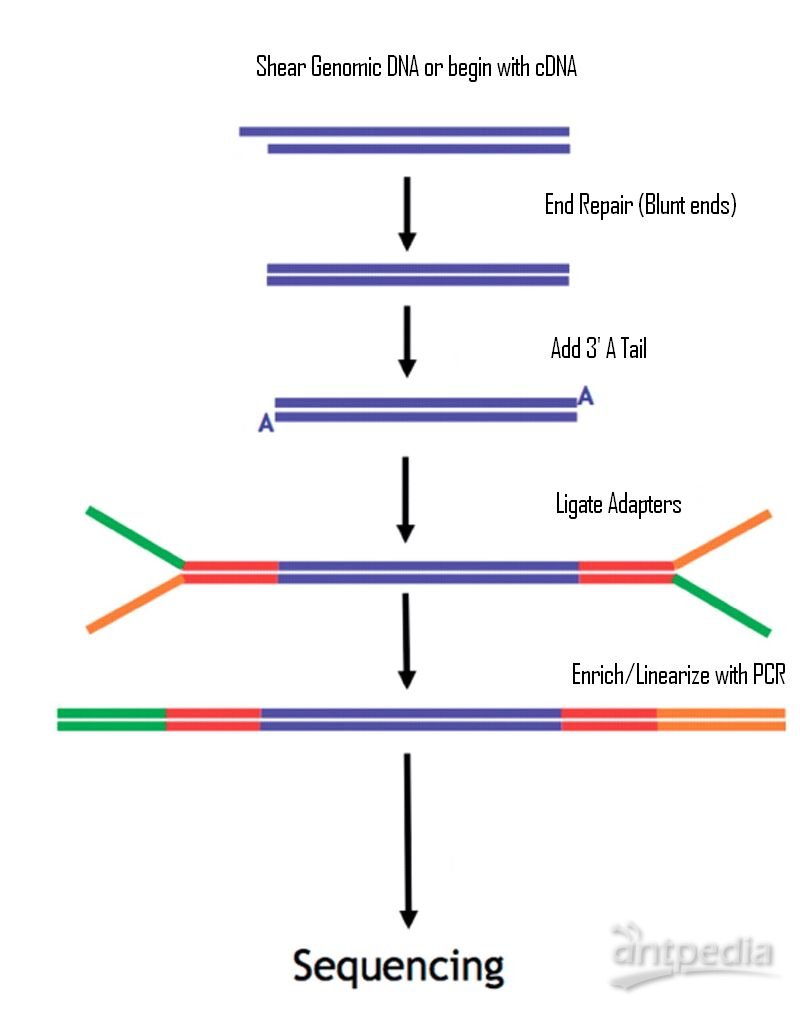
正如图所示,大概分为如下步骤。
用酶或者激光或者超声波将Genomic DNA或者由RNA反转组得到的双链cDNAs打断成小片段
打断是随机打断,有可能末端不平整,还需要用酶补平
补平之后,需要在3’端加A碱基
加上A之后,再加adapter
这时候,我们好像心里有那么点数,但是依然不知道adapter具体是怎么加上去的,也并不知道接头中,read1 sequncing primer, index, read2 sequencing primer,以及index sequencing primer到底在接头的什么地方。
那,是时候放出这张图了。
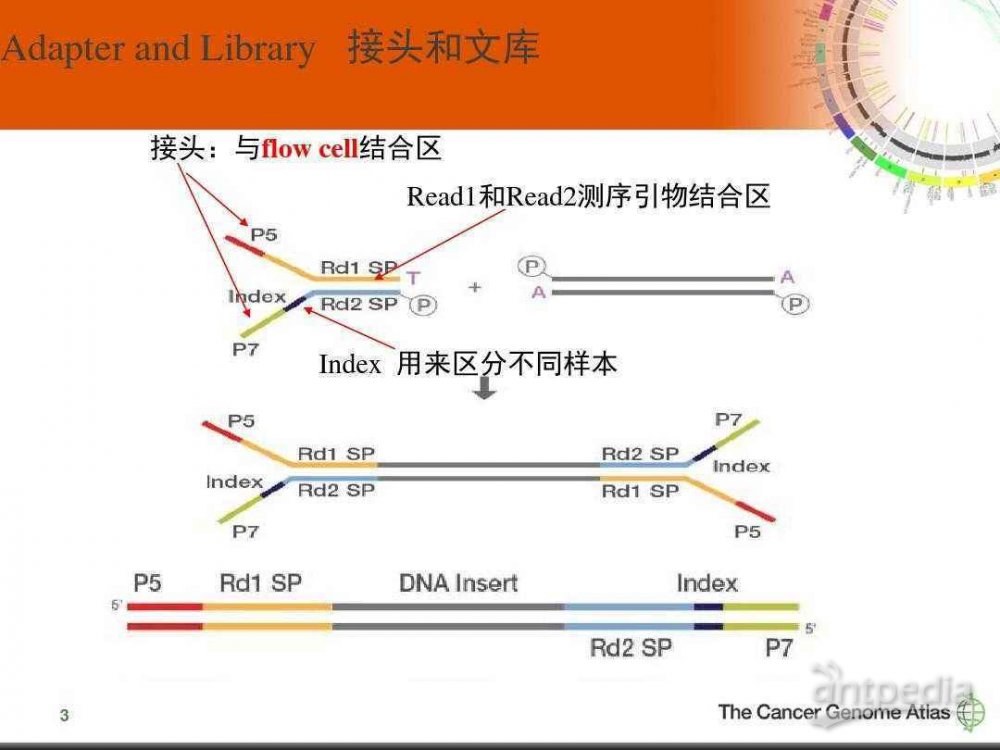
看完这张图,我们感觉对接头的添加这个过程的理解,好像多了几分。如果我们看上面这两张图,感觉就是在fragment DNA两端直接加了一个Y字形的引物,它被称人称为Full Y-adapter或者forked adapters。
但我们如果看illumina的官方视频,能够看到如下几帧介绍。

(上面的图是我从视频里截取下来,文字是根据我听到的加上去的)
从图片中我们能够看到,在“接头”添加之前,接头上好像已经有另一个叉形接头了,那这是咋回事呢? Y形接头不是直接添加到DNA fragment上的吗?
其实这是两种不同的indexing strategy导致的差异, 而这两种strategy的示意图,如下图所示。
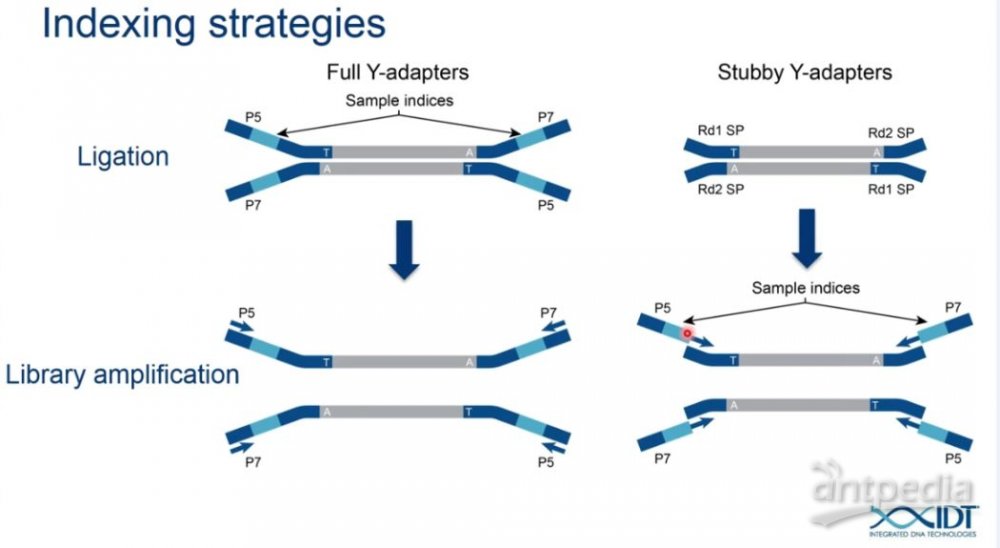
左边的是直接在fragment DNA的两端直接加上full Y-adapter, adapter中已经包括了和P5/P7 oligo互补的序列, index, 以及Read1/Read2的测序引物。
右边的那种是先在fragment DNA的两端加上PE adapter, 然后再引入和P5/P7 oligo互补配对的序列以及index序列。
一句话总结,这两种不同的indexing strategy的差别在于引入index序列的时机和方式不一样。
其实右边的图并不是画的特别形象,具体的的可以参看下面这张图,图片的来源是https://www.fimm.fi/en/services/technology-centre/sequencing/next-generation-sequencing/dna-library-preparation
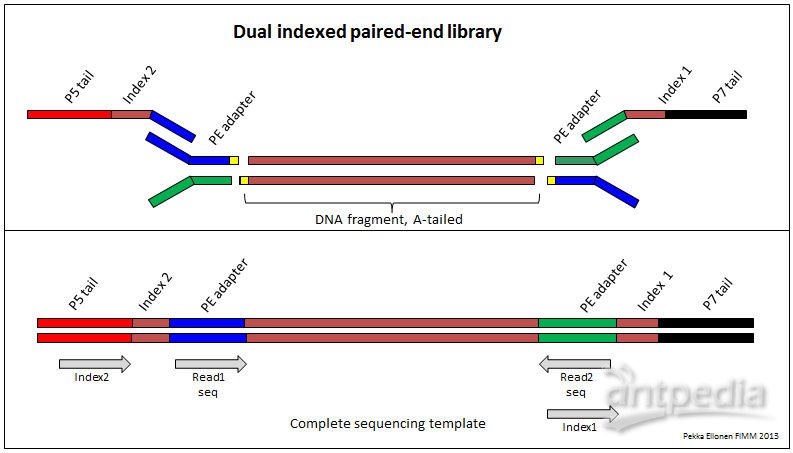
在这里我们能够清楚地看到,这种接头添加过程中,fragment DNA两端是先连上PE adapter, 然后再通过PCR引入的region complementary to P5/P7 sequence, index, and sequencing biding sites.
如果你的序列含有TruSeq Universal Adapter, 这时候可以采用如下去接头的代码。至于如何判断你的序列里到底有没有TruSeq Universal Adapter,可以下次单独写一篇来讲解。
去接头代码:
cutadapt --times 1 -e 0.1 -O 3 -m 30 -q 25,25 -u 8 -a AGATCGGAAGAGC -A AGATCGGAAGAGC -o trimmed.1.fastq.gz -p trimmed.2.fastq.gz reads.1.fastq.gz reads.2.fastq.gz
我们今天的收获是:
illumina文库构建的一般方式
illumina接头的两种添加方式(两种不同的indexing strategies)
如何用cutadapt去除TruSeq Universal Adapter
只是一个很小的细节,我们就讨论了这么多,今天就讨论到这里,下次我们再接着结合illumina官网的序列,用实实在在的碱基序列示意图来讲解,为什么要这么来去接头。
附上illumina官网的测序原理介绍视频如下:

