芝加哥大学汤玮欣团队(博士后肖玉兰为第一作者)在 Nature Biotechnology 期刊发表了题为:An adenine base editor variant expands context compatibility 的研究论文。
该论文通过巧妙的定向进化设计,从野生型大肠杆菌的TadA出发,经历五轮定向进化,获得了具有更高活性与更好序列兼容性的TadA8r,拓宽了腺嘌呤碱基编辑器(ABE)在疾病治疗的应用范围。
为了提高ABE在“RA”序列上的活性,研究团队构建了一套基于细菌抗生素抗性基因编辑的高通量筛选系统。研究团队将GATC序列置于抗生素抗性基因中,GATC的安装会导致抗生素抗性蛋白失活,只有当GATC序列被有活性的TadA*编辑为GGTC之后才能使抗生素抗性蛋白恢复活性。在E. coli中,几乎所有GATC中的A均被DNA腺嘌呤甲基转移酶 (Dam)在N6位甲基化。TadA*无法接受甲基化的A为底物, 仅有DNA复制过程中瞬时产生的未甲基化链可以成为TadA*的底物,即TadA*必须与Dam竞争底物。这种设计不仅迫使TadA*接受“RA”底物,同时还施加了更大的选择压力用于筛选具有更快脱氨酶活性的TadA*。从野生型TadA出发,经历五轮定向进化,研究团队获得了具有更高编辑活性与更好序列兼容性的TadA8r。
在体外动力学试验中,TadA8r在“GA”序列上的脱氨速率是目前两种活性最高突变体TadA8.20与TadA8e的9.5倍与11.1倍;在“TA”序列上的脱氨速率是TadA8.20与TadA8e的2.9倍与1.8倍。
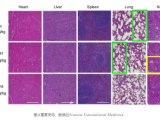
研究团队在约12000个疾病相关位点上系统性评估了ABE8r的编辑效率,编辑窗口,以及序列偏好性等。TadA8r向PAM远端拓宽了ABE8r的编辑窗口。除了在编辑“TA”序列时,ABE8r稍逊色于ABE8e之外,ABE8r在其他“CA”,“GA”以及“AA”序列上都表现出高于或者相当于其他ABEs的平均编辑效率。在纠正基因组中9407个与疾病相关的G到A突变位点时,ABE8r在其中41.9%的位点表现出优于ABE7.10, ABE8.20以及ABE8e的编辑效率。
TadA8r与其他CRISPR系统具有很好的兼容性,包括兼容具有更宽松PAM识别序列的其他SpCas9变种,SaCas9与其变种,LbCas12a及enAsCas12a等。在保持高的在靶编辑效率的同时,ABE8r还表现出显著低于ABE8e的DNA与 RNA水平脱靶效应。
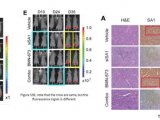
由于具有更宽的编辑窗口与更好的序列兼容性,ABE8r可以有效编辑其他ABEs难以编辑的疾病相关位点。例如在纠正导致Stargardt疾病的ABCA4 C.5882G>A (G1961E)突变时,ABE8r具有显著高于ABE7.10, ABE8.20以及ABE8e的编辑效率。
ABE8r作为具有更高活性与更好序列兼容性的腺嘌呤碱基编辑器,拓宽了ABE在疾病治疗的应用范围。由于TadA8.20已被成功应用于m6A检测, 凭借超高脱氨活性和更广泛的序列兼容性,TadA8r可能会加速用于核酸操作的复杂分子生物学工具的开发。