stLFR以其强大的虚拟隔离共标记技术(Virtual Co-Barcoding),通过单管操作就能轻松的获得基因组长片段信息。为了能高效的利用庞大的barcode信息,华大智造生信开发团队针对stLFR数据结构特点开发一款全新、自动化的stLFR数据分析工具。免费获取方式为:https://github.com/MGI-tech-bioinformatics/stLFR_v1.1 。
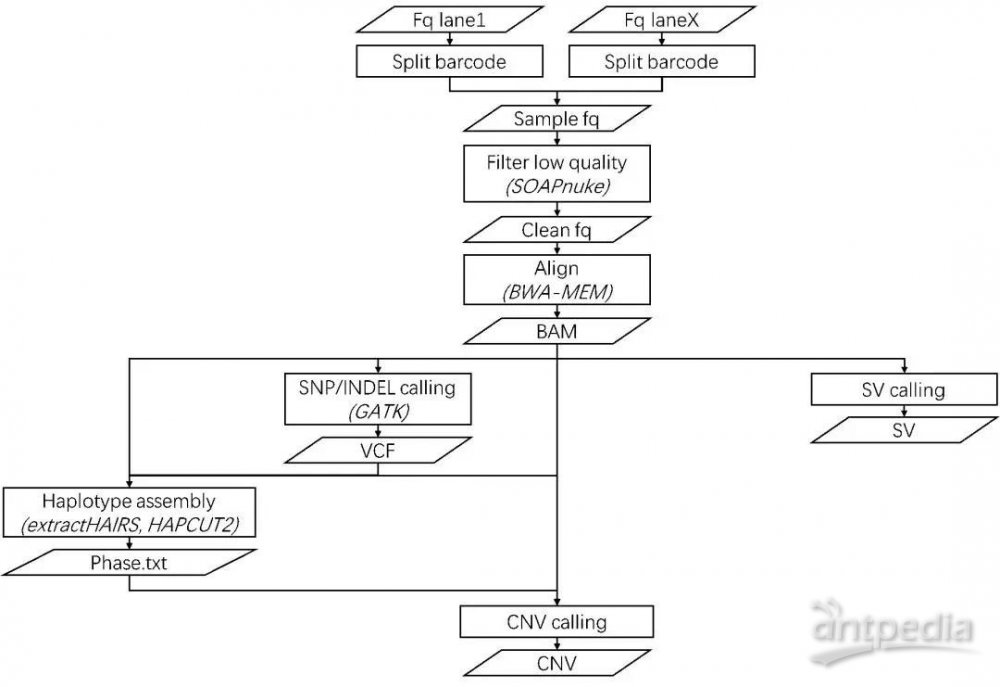
图1. stLFR数据分析工具示意图。
(从下机数据开始,依次进行barcode拆分、低质量数据过滤、基因组比对、SNP/INDEL检测、单倍体组装、CNV检测和SV检测,最终得到stLFR数据报告)
数据展示
通过对2个NA12878数据结果来介绍stLFR数据分析工具的报告内容,详细展示工具能提供什么结果。
Co-Barcode聚类分析
利用DNA分子共标签技术,华大智造stLFR文库制备试剂盒能获取较好的长片段文库。那么在实际stLFR数据中的表现将通过三个图表展示。

图2. stLFR数据的barcode分布、覆盖。
每个barcode连接的片段数目(a, b)、每个片段的覆盖度(c, d)、每个片段的长度分布(e, f)
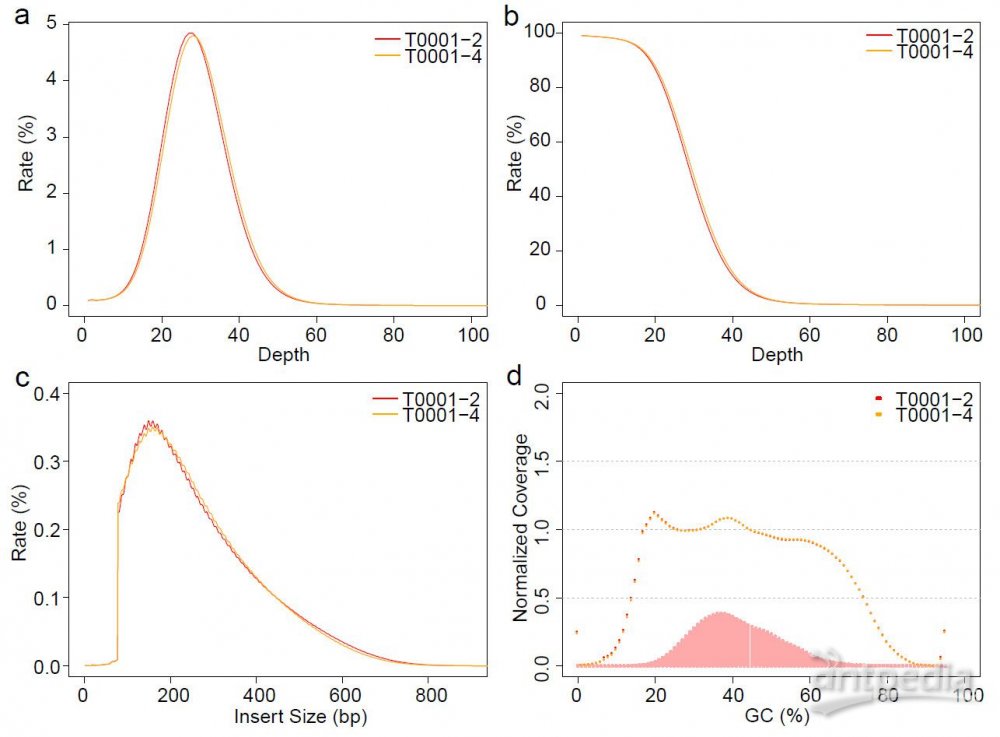
图3. stLFR数据深度覆盖、插入片段、GC bias示意图。
stLFR数据深度分布图(a)、累积深度分布图(b)、插入片段分布图(c)及GC bias示意图(d)。
单倍体组装
作为stLFR技术的重点之一,工具使用HapCUT2软件进行单倍体组装分析,得到较好的基因组组装结果。两个stLFR样品的单倍体组装的最大N50达到15M,phasing rate在99%+。
表1. stLFR数据单倍体组装统计
|
染色体 |
T0001-2 |
T0001-4 |
||||
|
Switch rate |
N50 |
Phasing rate |
Switch rate |
N50 |
Phasing rate |
|
|
1 |
0.0098 |
6,012,525 |
99.82% |
0.0099 |
7,203,376 |
99.83% |
|
2 |
0.0004 |
8,066,973 |
99.87% |
0.0004 |
12,434,593 |
99.85% |
|
3 |
0.0005 |
8,618,950 |
99.90% |
0.0005 |
6,233,405 |
99.90% |
|
4 |
0.0104 |
5,729,259 |
99.88% |
0.0105 |
9,066,777 |
99.87% |
|
5 |
0.0098 |
6,725,647 |
99.90% |
0.0099 |
9,571,060 |
99.89% |
|
6 |
0.0106 |
7,644,767 |
99.90% |
0.0107 |
8,810,819 |
99.88% |
|
7 |
0.0038 |
6,663,634 |
99.87% |
0.0038 |
7,858,180 |
99.85% |
|
8 |
0.0092 |
5,189,893 |
99.88% |
0.0093 |
6,643,768 |
99.87% |
|
9 |
0.0100 |
8,905,613 |
99.73% |
0.0099 |
7,147,435 |
99.67% |
|
10 |
0.0046 |
9,189,953 |
99.89% |
0.0045 |
10,304,910 |
99.88% |
|
11 |
0.0109 |
9,111,332 |
99.88% |
0.0111 |
9,650,259 |
99.90% |
|
12 |
0.0005 |
6,031,100 |
99.87% |
0.0003 |
9,561,399 |
99.89% |
|
13 |
0.0005 |
9,928,540 |
99.92% |
0.0005 |
11,438,457 |
99.90% |
|
14 |
0.0006 |
5,249,922 |
99.84% |
0.0005 |
7,533,475 |
99.88% |
|
15 |
0.0008 |
5,245,452 |
99.78% |
0.0007 |
6,299,999 |
99.73% |
|
16 |
0.0004 |
10,946,109 |
99.85% |
0.0005 |
13,070,565 |
99.88% |
|
17 |
0.0005 |
4,343,525 |
99.83% |
0.0004 |
4,343,525 |
99.82% |
|
18 |
0.0093 |
7,155,753 |
99.93% |
0.0094 |
12,886,313 |
99.93% |
|
19 |
0.0138 |
7,781,865 |
99.93% |
0.0139 |
15,193,578 |
99.89% |
|
20 |
0.0004 |
13,475,627 |
99.93% |
0.0003 |
7,030,408 |
99.92% |
|
21 |
0.0004 |
7,205,559 |
99.85% |
0.0004 |
9,724,771 |
99.88% |
|
22 |
0.0111 |
10,422,522 |
99.81% |
0.0109 |
13,163,868 |
99.86% |
|
X |
0.0007 |
2,506,710 |
99.67% |
0.0008 |
3,069,697 |
99.64% |
|
Genome |
0.0056 |
6,653,785 |
99.86% |
0.0056 |
7,766,036 |
99.85% |
全基因组范围N50达到7.8M,phaseing rate在99.8%。
CNV、SV检测
利用分子标签和长片段信息,stLFR可以对多种结构变异进行准确检测。华大智造stLFR数据分析工具中使用全新的CNV、SV分析软件进行结构变异检测。
表2. stLFR数据的CNV检测结果
|
Sample |
T0001-2 |
T0001-4 |
|
Total_SNP |
3,751,967 |
3,751,478 |
|
dbSNP_rate |
99.40% |
99.40% |
|
Novel_SNP |
22,683 |
22,450 |
|
Novel_SNP_Rate |
0.60% |
0.60% |
|
Ti/Tv |
2.04 |
2.04 |
|
Total_INDEL |
855,874 |
853,589 |
|
dbINDEL_Rate |
84.97% |
85.15% |
因展示数据是NA12878样品,因此使用NA12878的变异标准集进行变异评估。
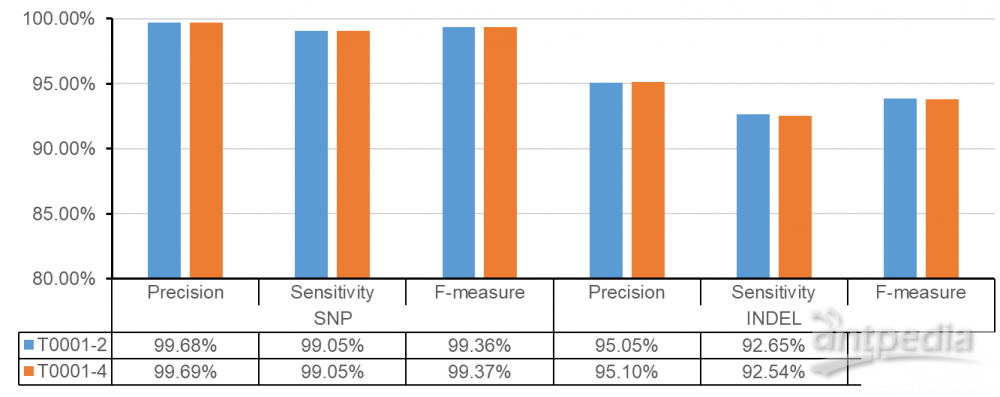
两个NA12878 stLFR样品的SNP Sensitivity和Precision都达到99%+,INDEL的Sensitivity也在92%+。
基本数据统计
以表格展示了样品的数据信息,包括数据量、stLFR文库中barcode组合及实际数据中barcode拆分统计、拆分后有效数据的比对率、重复率、深度、基因组覆盖度、平均插入片段等关键信息;以图形展示了数据深度分布、GC bias、插入片段等数据信息(见图3)。
表5. stLFR流程基本数据统计。
<table border="1" width="550" cellspacing="0" cellpadding="0" ">
|
Sample name |
T0001-2 |
T0001-4 |
|
Total barcode type |
3,623,878,656 |
3,623,878,656 |
|
Barcode number |
51,646,936 |
52,700,541 |
|
Barcode type rate |
1.43% |
1.45% |
|
Reads pair number |
794,472,074 |
786,248,806 |
|
Reads pair number(after split) |
689,581,728 |
679,315,099 |
|
Barcode split rate |
86.80% |
86.40% |
|
Mapping rate |
99.82% |
99.84% |
|
Paired mapping rate |
99.43% |
99.44% |
|
Mismatch rate |
0.55% |
0.59% |
|
Duplicate rate |
34.66% |
29.79% |
|
Total depth |
52.96 |
52.42 |
|
Split barcode(G) |
137.92 |
135.86 |
|
Dup depth |
30.04 |
31.8 |
|
Average sequencing depth |
29.77 |
30.3 |
|
Coverage |
99.09% |
99.08% |
|
Coverage at least 4X |
98.79% |
98.77% |
|
Coverage at least 10X |
97.94% |
97.93% |
|
Coverage at least 20X |
86.73% |
87.90% |
|
Mean insert size |
268.35 |
265.64 |
运行资源
为说明需要,运行资源以30X人类基因组数据为标准。stLFR数据分析工具从原始数据输入开始,经过总计9个处理模块得到结果,分析处理一个30X数据量的stLFR样本总计需要2.5天,全程最大内存消耗为SV模块的60G,最大存储消耗为低质量数据过滤时的103G,最大时间消耗为比对去重模块的24.9小时,更具体的信息可看下方表格。
表6. stLFR数据分析工具运行资源。
|
步骤 |
最大内存(G) |
最大存储(G) |
时间(h) |
|
fq_BarcodeSplit |
15 |
111 |
6 |
|
fq_Filter |
4 |
103 |
9.5 |
|
fq_AlignSortMarkdup |
10 |
60 |
24.9 |
|
bam_Split |
1 |
60 |
0.7 |
|
bam_GATK |
10 |
1.3 |
5.8 |
|
bamvcf_HaplotypeAssembly |
15 |
2.8 |
2.5 |
|
bamvcfhap_CNV |
15 |
10 |
2.6 |
|
bam_SV |
60 |
10 |
5 |
|
report_Stat |
8 |
0.1 |
2.6 |
|
总计 |
- |
307 |
59.6 |
处理约30X人类基因组数据量的stLFR样品需要总计约307G硬盘存储、60G运算内存、2.5天时间。
运行命令
stLFR数据分析工具需在Linux环境运行,自Github下载获取软件并解压后可以取得直接运行的运行程序stLFR,直接运行程序可以查看帮助信息。
1. stLFR
2. Usage:
3. perl stLFR [options]
4.
5. Arguments:
6. sample.list
7. List of input.
8.
9. Format: "sample path [ barcode ]"
10.
11. If one sample have 2 lanes of fastq, there should be two lines in the fqlist file for this sample.
12. There are at least 2 columns separated by blank(s) or tab(s) in each line:
13. the 1st column is sample name, no blank or chinese character, required
14. the 2nd column is the lane path of fastq files, must contain *_1.fq.fqStat.txt, required
15. the 3rd column is the barcode positions [ 101_10,117_10,133_10 ]
16.
17. Options:
18. --outdir
19. Output path. [./]
20.
21. --ref
22. Human reference version . [hs37d5]
23.
24. --cpu <70>
25. CPU number. [70]
26.
27. --help|-h
28. Print this information.
主程序必要参数只有一个,即样品信息列表(sample.list),其中以最少2列的形式写明样品及下机数据路径。
软件列表
|
软件 |
版本 |
|
Linux |
不限 |
|
Perl |
V5.26.1 |
|
SOAPnuke |
1.5.6 |
|
BWA |
0.7.17-r1198-dirty |
|
samtools |
1.3 (using htslib 1.3) |
|
java |
1.8.0_101 |
|
Picard |
2.0.1 |
|
GATK4 |
4.0.3.0 |
|
bgzip |
1.5 |
|
tabix |
1.3.1 |
|
python |
2.7.14 |
|
bcftools |
1.2 |
|
HAPCUT2 |
- |
|
python3 |
3.6.3 |
光看不过瘾,下载Demo数据尝试一下吧
以上演示的demo数据已上传CNSA,可免费下载:T0001-2和T0001-4:
|
样品 |
T0001-2 |
T0001-4 |
||
|
Slide |
V300014293B |
|||
|
Lane |
L01 |
L02 |
L03 |
L04 |
|
物种 |
NA12878 |
|||
|
建库方法 |
stLFR |
|||
|
试剂盒版本 |
MGIEasy stLFR 文库制备试剂盒V1.0 |
|||
|
测序仪 |
MGISEQ-2000 |
|||
|
测序时间 |
2019-01-29 |
|||
|
测序模式 |
PE100 |
|||
|
数据量(G) |
78.70 |
80.19 |
79.02 |
78.23 |
|
Q20(%) |
91.92 |
92.09 |
91.46 |
91.67 |
|
Q30(%) |
81.33 |
81.78 |
80.70 |
81.03 |
|
GC(%) |
42.70 |
42.70 |
42.90 |
42.80 |
|
CNSA编号 |
CNX0045104 |
CNX0045105 |
CNX0058543 |
CNX0058544 |
以上演示的2个demo数据产自MGISEQ-2000平台,数据已上传至CNSA,可免费下载。
T0001-2:ftp://ftp.cngb.org/pub/CNSA/CNP0000387/CNS0057111/
T0001-4:ftp://ftp.cngb.org/pub/CNSA/CNP0000387/CNS0094773/
参考文献
[1] Wang O et al: Efficient and unique cobarcoding of second-generation sequencing reads from long DNA molecules enabling cost-effective and accurate sequencing, haplotyping, and de novo assembly. Genome Res 2019, 29(5):798-808.