师 兄:师妹,怎么了,看你这两天都不开心的样子?
师 妹:愁啊,文章还没写完,下个月就要送审了。之前做了转录组和蛋白组分析,可是不知道怎么把它们联合深入挖掘,总感觉差那么一点点。
师 兄:别急别急,好像前几天有销售过来说,欧易生物的组学联合分析叒升级了,这个公司很靠谱,之前咱们老板跟他们合作过的,专业技术过硬,售后应答反馈及时,而且作图结果可直接用于文章发表。
师 妹:哇,那我有救了!我和欧易生物的销售联系下~
组学联合分析
大升级
下面就和我们的小师妹一起来看看这次升级的内容吧~!
流程图
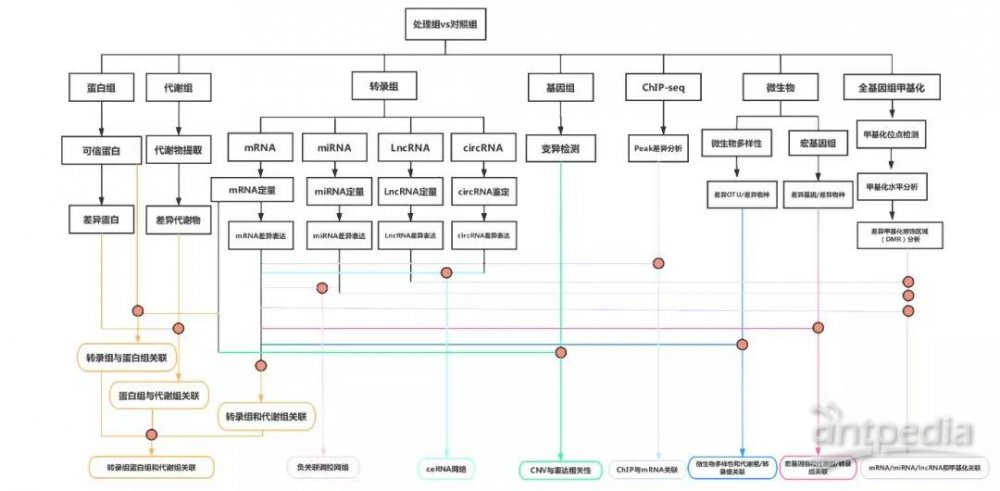
点击查看大图
本次升级主要体现在以下几个方面:
一、联合分析覆盖范围更为广泛
1. 增加ChIP-seq和表达谱联合分析
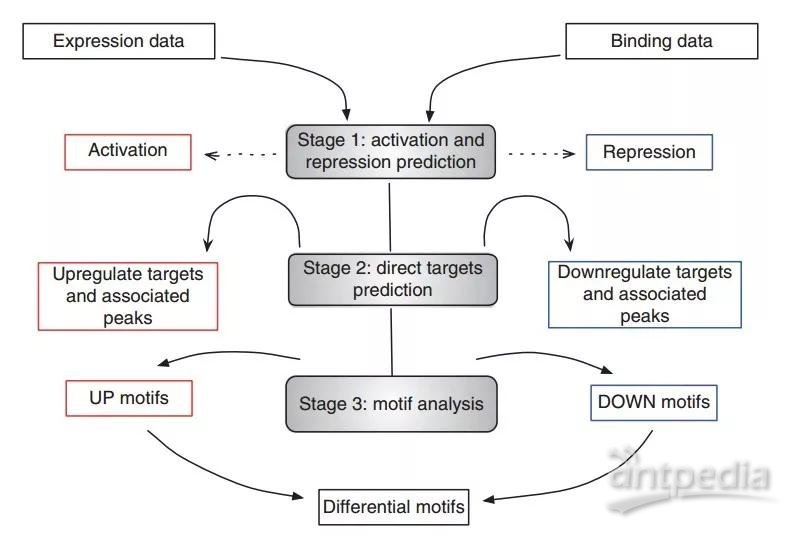
1.1.1 BETA软件分析流程[1]
图为BETA软件分析流程。第一步:分析差异表达数据和ChIP-seq结合数据,来预测因子激活或抑制基因的表达情况。第二步:通过上调或下调预测直接靶基因。第三步:进行Motif分析以识别促进上调或下调的辅因子。
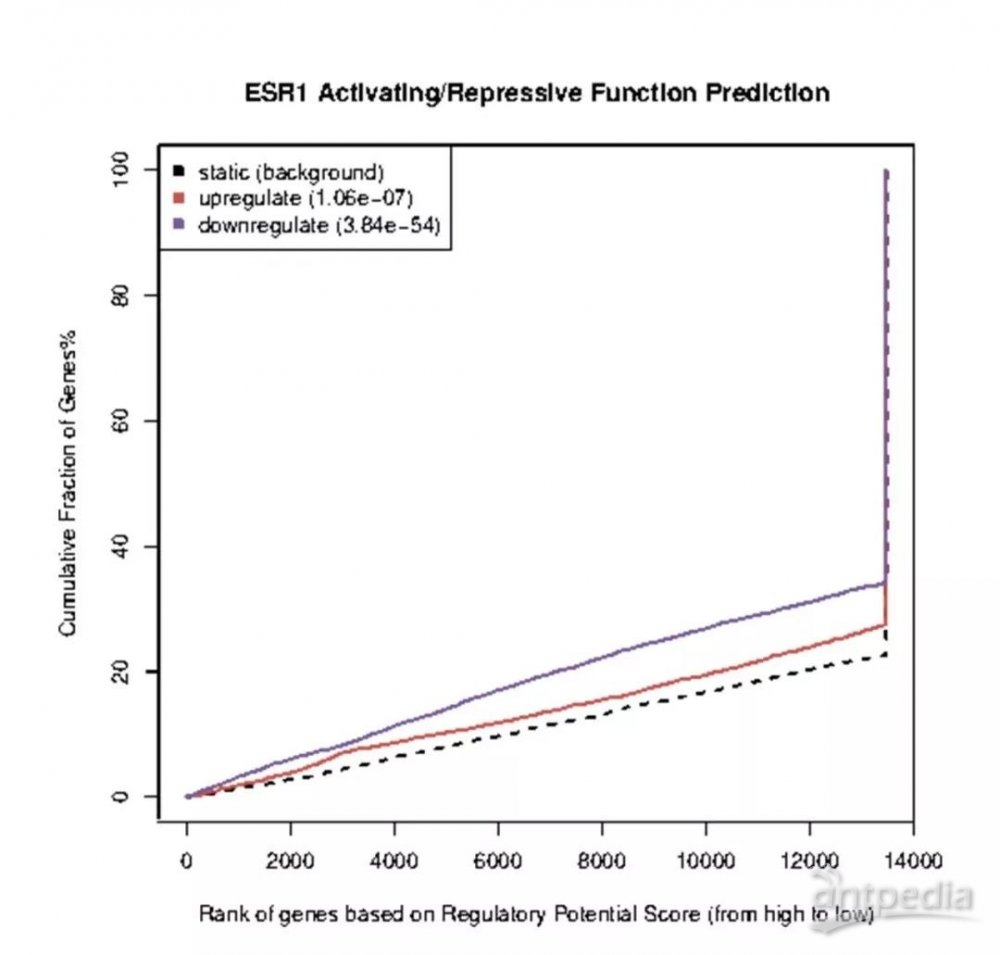
1.1.2转录因子或者染色质调节因子的调控潜能预测
图中红色及紫色曲线分别代表上调基因、下调基因;黑色虚线代表未差异表达基因,作为背景。其中横轴代表调控潜能从高到低排列累积的基因数目;纵轴为基因累计比率。同时通过KS-检验(Kolmogorov-Smirnov test)来计算上调基因,下调基因与未差异表达基因的差异显著性,pvalue值见图注中upregulate及downregulate后标注数值。若上调基因累计比率显著高于下调基因以及背景,说明该转录因子或者染色质调节因子具有激活功能;若下调基因累计比率显著高于上调基因以及背景,说明该转录因子或者染色质调节因子具有抑制功能;若上调基因累计比率与下调基因累计比率相当,且显著高于背景,说明该转录因子或者染色质调节因子同时具有激活和抑制功能。
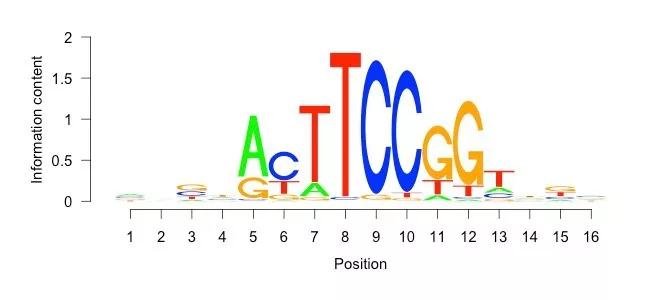
1.1.3 Motif分布示意图
2. 增加lncRNA/miRNA和甲基化(WGBS)联合分析
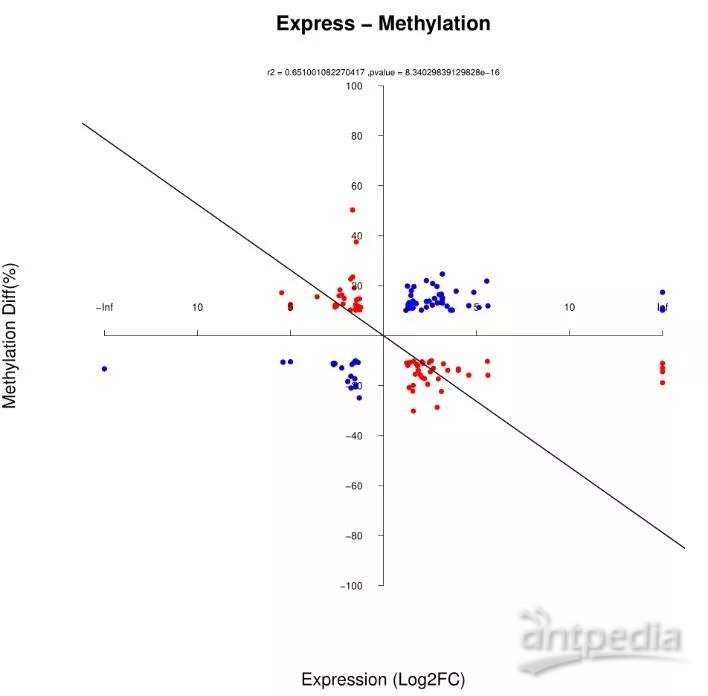
1.2.1 差异基因和甲基化四象限图
图中红色表示差异表达RNA的FoldChange在2倍以上的负相关位点,蓝色表示差异表达RNA的FoldChange在2倍以上正相关的位点。
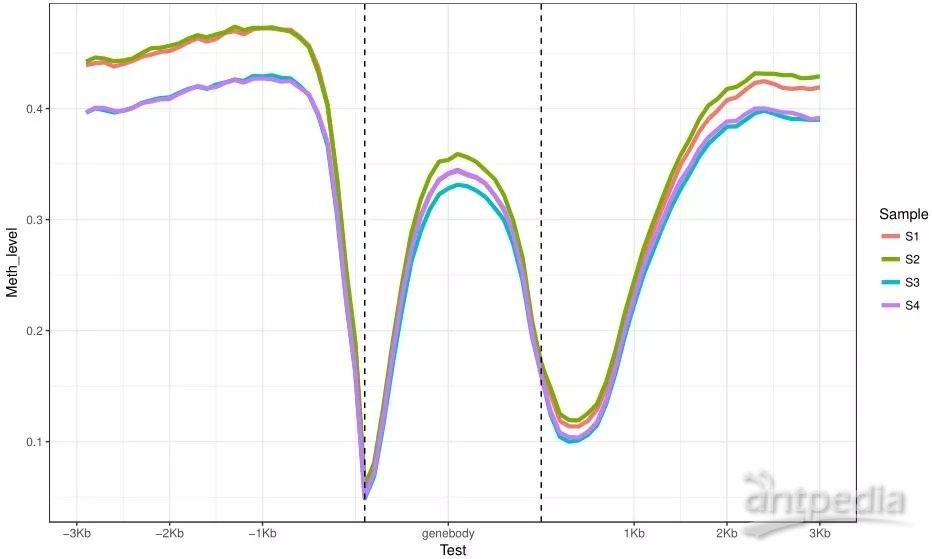
1.2.2 mRNA/lncRNA/miRNA 上下游甲基化水平的profiling分析
图中横坐标为基因区域,纵坐标表示甲基化水平。
3. 增加CNV和表达谱联合分析
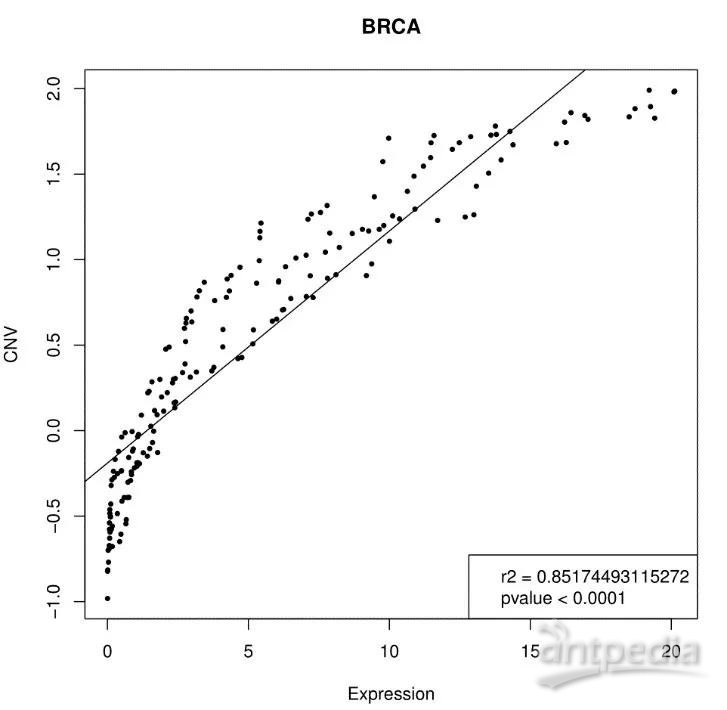
1.3.1 CNV和表达量关联图
图中横坐标为基因表达量,纵坐标为CNV拷贝数。
二、已有联合分析模块内容更为丰富
1.转录组和蛋白组关联
(1)差异基因和蛋白的相关性分析
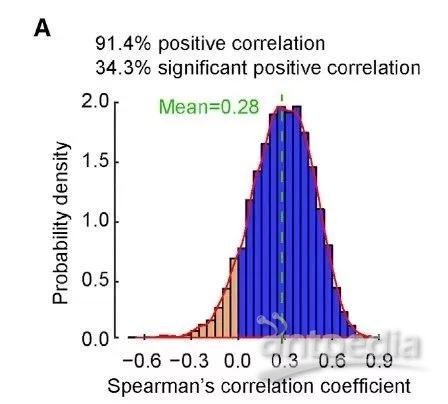
2.1.1.1差异基因和蛋白相关性分析[2]
图中展示的是在差异基因和蛋白一一对应关系下,对所有相关性的结果进行绘图的结果。横轴为相关性系数数值范围,纵轴为相关性系数密度分布。每一个柱子代表在对应相关性系数范围内的关系对的分布,柱子越高代表在该系数范围内的概率密度越大,即关系对越多。同时展示了密度分布曲线图,曲线图越高则对应的概率密度越大。黄色代表负相关,蓝色代表正相关。
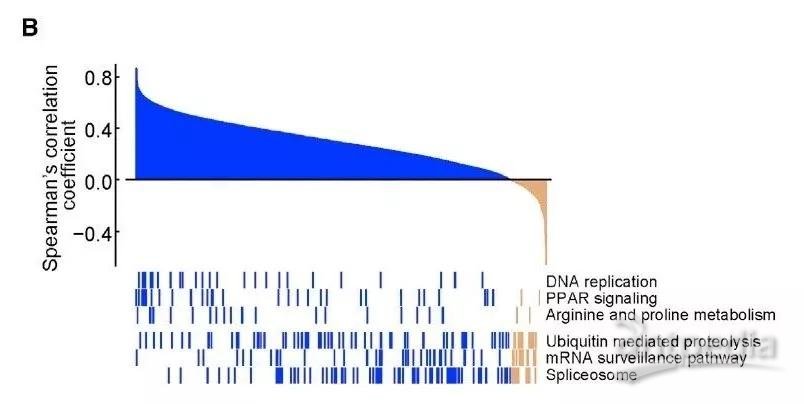
2.1.1.2 差异基因和蛋白KEGG注释前六个 pathway占比及其相关性[2]
图中对KEGG 注释条目最多的前6个pathway涉及到的基因和蛋白关系进行热图的绘制。图形上部分为涉及到6个pathway的所有基因和蛋白关系对相关性,黄色代表负相关,蓝色代表正相关。横轴代表转录和蛋白对应的关系对。图形下方为对应的关系对在6个pathway的分布,白色代表该pathway中并未找到对应关系对,黄色代表负相关,蓝色代表正相关。
(2)转录组与蛋白组GO Term 分析
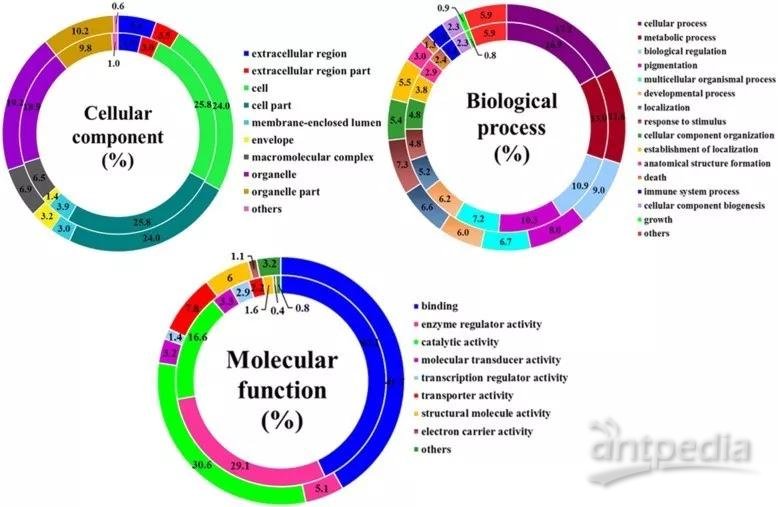
2.1.2.1差异蛋白和差异mRNA GO条目对比示意图[3]
图中不同颜色分别为不同层级的GO条目及相应的占比,内环为差异mRNA的分布,外环为差异蛋白的分布。
2.微生物和代谢组/转录组联合分析
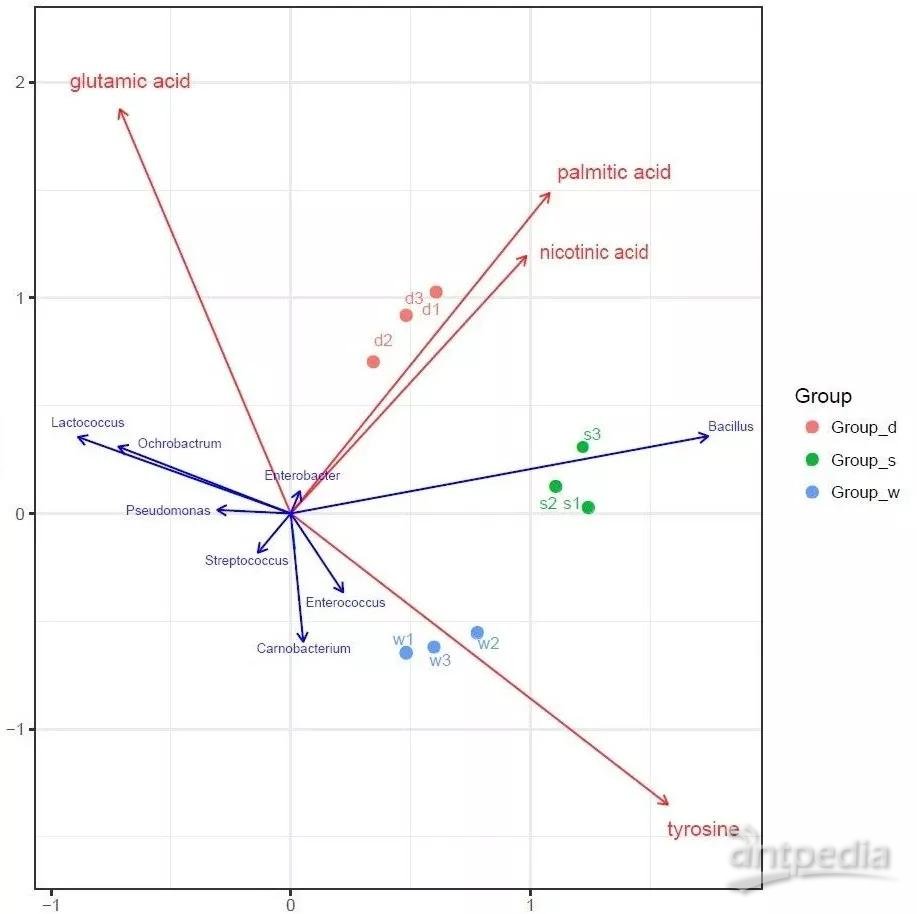
2.2.1 差异微生物与差异代谢物CCA分析
图中红色箭头代表不同的基因/代谢物,蓝色箭头代表不同物种。基因/代谢物之间的夹角为锐角时表示微生物与基因/代谢物之间呈正相关,钝角时呈负相关。物种与基因/代谢物之间的夹角代表物种与基因/代谢物间的正、负相关关系(锐角:正相关;钝角:负相关;直角:无相关性)基因/代谢物的射线越长,说明该基因/代谢物的影响程度越大。
3.Pathway分析增加color key
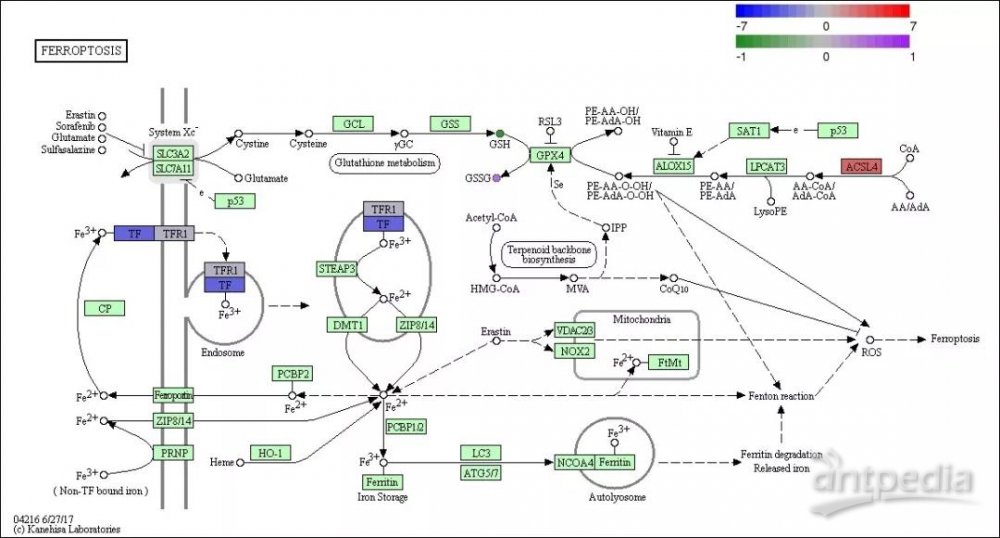
2.3.1 差异基因与差异代谢物pathway 图
图中方框代表基因,圆圈代表代谢物;颜色以红蓝渐变来显示基因的具体表达情况,红色表示上调,蓝色表示下调,以绿紫渐变来显示代谢物的具体表达情况,紫色表示上调,绿色表示下调。
4.转录/蛋白和代谢组增加KGML网络分析
KGML文件是KEGG数据库中的一个子库,既包含KEGG pathway中图形对象的关系,也包含了KEGG GENES数据库中直系同源基因的信息。通过这些信息可以得到基因产物和代谢物的网络关系,方便更加系统地研究转录组、蛋白组和代谢组之间的相互作用。
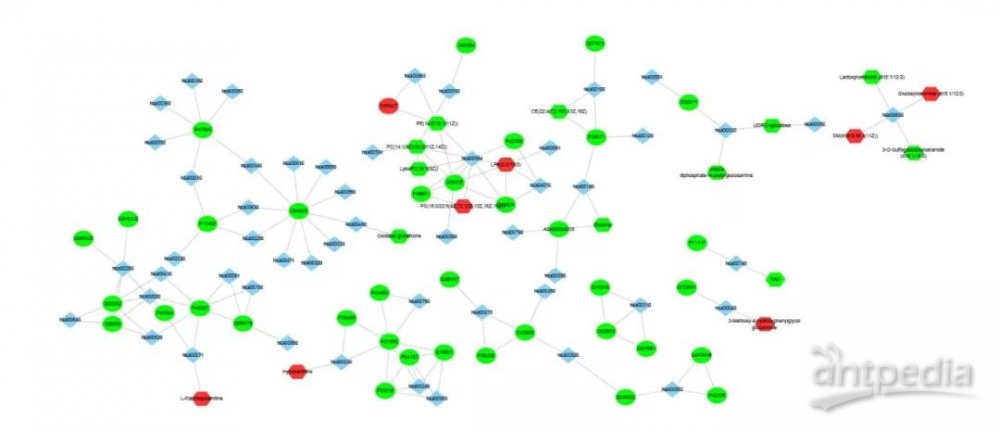
2.4.1差异基因、蛋白和代谢物 KGML 网络图
图中圆圈表示基因或基因产物,六边形表示代谢物,菱形表示通路名称。红色表示上调的基因、基因产物或者代谢物,绿色表示下调的基因、基因产物或者代谢物 。
5.ceRNA分析重新优化
由于原有的ceRNA[4-7] 分析算法参考文章对应的网站难以正常访问,所以对算法进行了替换,更改为MuTATE算法,该算法来源于cell杂志(Tay Y .etc 2011)[8],主要强调了ceRNA结合的以下几个方面:
(1)mRNA上共享miRNA数量的多少;
(2)靶基因上每个miRNA 的MREs数量以及这些MREs的分布跨度;
(3)靶基因上每个miRNA的MREs分布密度;
(4)靶基因上MREs与miRNA数量的比值。
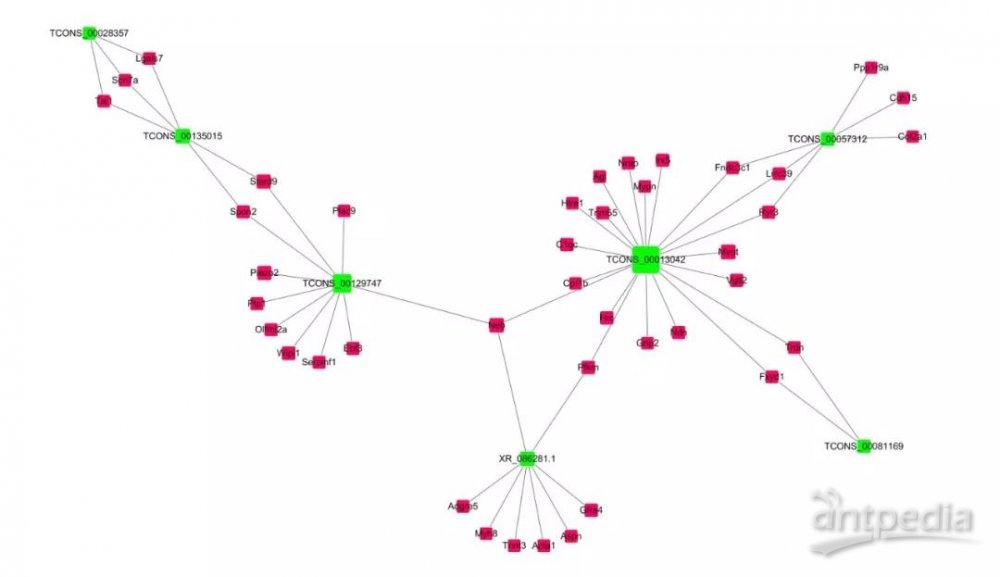
2.5.1 ceRNA调控关联网络图
图中红色为基因,绿色为lncRNA或者circRNA ,连线代表ceRNA关系对。
6. miRNA-转录组负调控关联分析
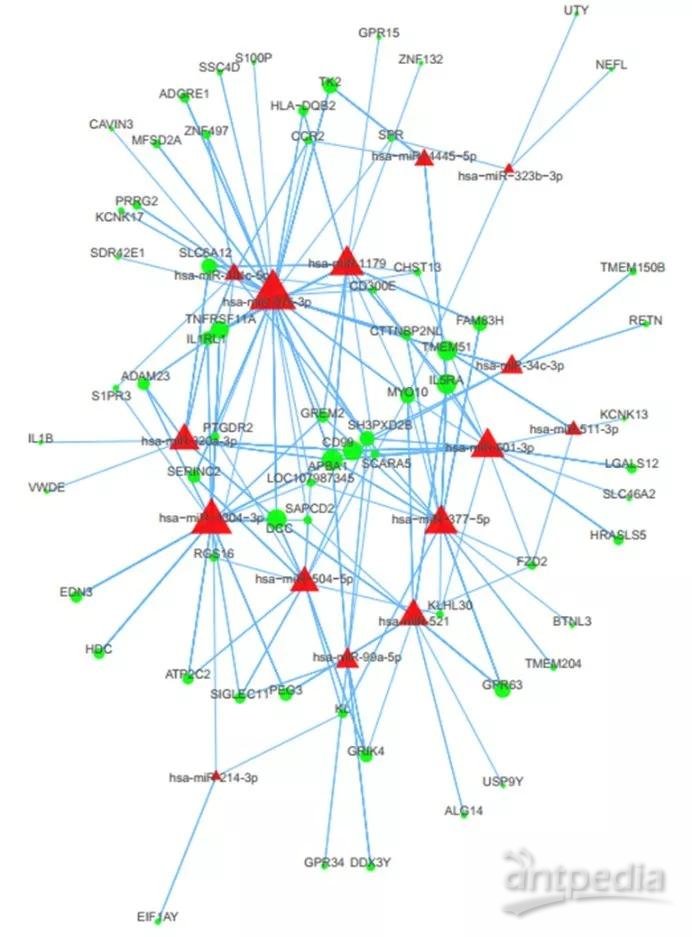
2.6.1 miRNA与mRNA/circRNA/lncRNA负调控关联网络图
图中miRNA为三角形,mRNA/circRNA/lncRNA为圆形,红色代表上调,绿色代表下调,图形越大说明与之相连的节点越多。
三、报告可读性增强
1.完善了所有表格和图片说明,报告文字描述及表头解释更加深入浅出。
2.可视化更美观,每部分增加了流程图,分析逻辑更加清晰。
3.原有word报告文档升级成网页版文档,视觉体验更加精彩。
看到这里,相信大家对欧易的多组学联合分析升级有了初步了解,感兴趣或有分析需求的老师,欢迎咨询当地销售~
文末彩蛋:围绕单细胞的多组学分析也在紧锣密鼓地进行,具体升级内容稍后揭晓,敬请期待!
参考文献
1. Su Wang, Hanfei Sun, Jian Ma, et al. Chongzhi Zang Target analysis by integration of transcriptome and ChIP-seq data with BETA[J]. Nature protocols, 2502-2515(2013).
2. Mun D, Bhin J,Kim S, et al. Proteogenomic Characterization of Human Early-Onset Gastric Cancer[J].Cancer Cell 35, 111–124(2019)
3. Wenting Dai, Quanjuan Wang, Fengqi Zhao, et al. Understanding the regulatory mechanisms of milk production using integrative transcriptomic and proteomic analyses: improving inefficient utilization of crop byproducts as forage in dairy industry. BMC Genomics,19:403 (2018)
4. Enright AJ, John B, Gaul U, et al. MicroRNA targets in Drosophila.Genome Biol5, doi: 10.1186/gb-2003-5-1-r1 (2003).
5. Das S, Ghosal S, Sen R, et al. lnCeDB: database of human long noncoding RNA acting as competing endogenous RNA.Plos one 9, e98965 (2014).
6. Liu K, Yan Z, Li Y, et al. Linc2GO: a human LincRNA function annotation resource based on ceRNA hypothesis.Bioinformatics 29, 2221-2222 (2013).
7. Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language .Cell 146, 353-358 (2011).
8. Tay Y, Kats L, Salmena L, et al. Coding-independent regulation of the tumor suppressor PTEN by competing endogenous mRNAs. Cell 147, 344-357 (2011).
#END#
本文系欧易生物原创
转载请注明本文转自欧易生物