细菌和古生菌构成了生命世界的绝大部分,但是它们中的绝大部分物种,包括与人类有密切关联的物种,从未被分离或培养过。
对来自自然微生物群体的DNA进行测序已可以鉴定以前未知的分类群(taxa),在某些情况下还提供了有关这些有机体的详细基因组信息。然而,美国加州大学圣地亚哥分校微生物学家Karsten Zengler说,拥有序列数据就像“拥有机器的零件清单”。这“并不能告诉你这台机器将做什么。”
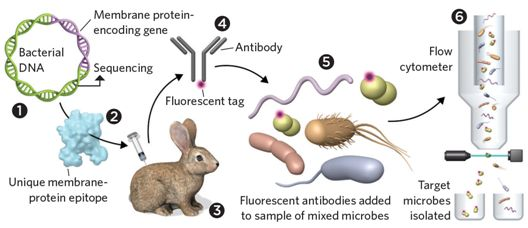
为了更好地了解微生物的生理学和功能,科学家们需要研究活的样本,或至少研究整个细胞。为此,在一项新的研究中,美国橡树岭国家实验室微生物遗传学家Mircea Podar及其同事们研究了未培养微生物(uncultured microbe, 即不能在实验室培养的微生物)的序列数据,以便设计工具来捕获这些微生物。相关研究结果近期发表在Nature Biotechnology期刊上,论文标题为“Targeted isolation and cultivation of uncultivated bacteria by reverse genomics”。
图片来自Nature Biotechnology, 2019, doi:10.1038/s41587-019-0260-6。
通过着重关注存在于人类口腔中的细菌,Podar及其研究团队将来自未培养微生物的可用序列数据与先前可培养细菌(cultured bacteria)的序列进行比较,以鉴定潜在的细胞表面蛋白。随后,他们利用潜在的细胞表面蛋白尽可能地选择未培养微生物特有的肽片段,并将这些肽注射到兔子中以产生针对它们的抗体,利用荧光标记物标记这些抗体。将这些荧光标记抗体添加到来自人类唾液的微生物样本中,就可检测出具有相应表面蛋白的细菌,并通过荧光激活细胞分选术(FACS)分离出它们。
Podar团队分离出两种不同的以前不能培养的口腔细菌:TM7和SR1。他们通过使用精心选择的培养基来培养这两种口腔细菌。除了富集这两种靶细菌本身外,这种抗体介导的技术还提取出与TM7和SR1存在物理关联性的特定类型的细菌。实际上,对TM7和SR1的成功培养的部分原因可能是共分离它们的生长所需的其他微生物。
Podar说,虽然原则上这种技术可能可应用于任何细菌,但是对每种新的靶菌株,人们必须仔细设计用于产生抗体的肽。这种设计是在与其他微生物的已知表面蛋白的相似性与对靶微生物的潜在表面蛋白的特异性之间保持一种谨慎的平衡。因此,他说,“它永远不会成为工具包。”相反,“这是一种可以增加你成功机会的指导性方法。”
利用这种称为反向遗传学(reverse genetics)的技术,科学家们可以获得特异性的抗体来捕获以前不可培养的微生物。Zengler(未参与这项研究)说,这种技术“将帮助我们获得更多的可培养微生物,并且更多地了解我们仅从基因组指纹中了解的微生物的信息”。

