![]() 型号:Sequenom MassArray
型号:Sequenom MassArray
![]() 品牌:欧易生物
品牌:欧易生物
![]() 产地:上海
产地:上海
Sequenom MassArray
Sequenom® MassArray甲基化研究平台适合于特定DNA片段甲基化的研究。在经过全基因组甲基化芯片或启动子区甲基化芯片初步筛选后,对差异甲基化基因位点的技术验证和生物学独立验证;或者研究者已确立甲基化基因目标,或只对某些特定DNA甲基化的检测有兴趣的情形。Sequenom ® MassArray DNA甲基化采用专业的引物设计软件,无需进行PCR产物纯化,无需任何荧光标记,是研究几个到几百个甲基化位点的理想手段。
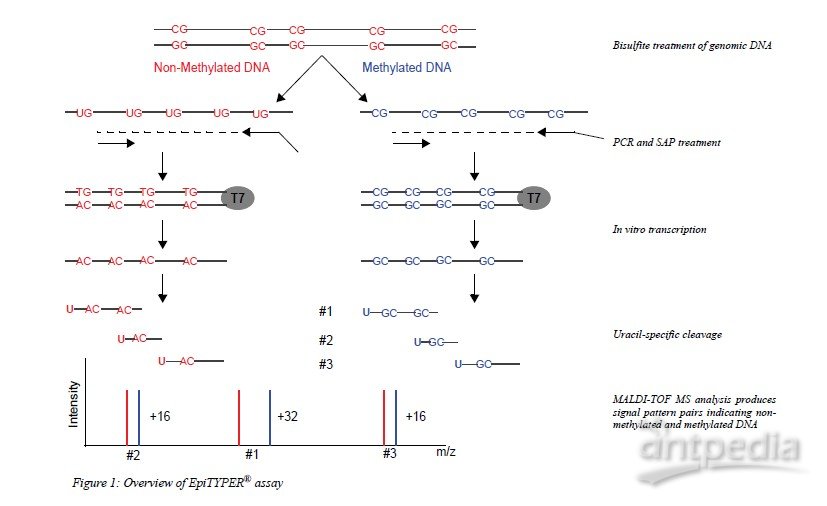
实验步骤
1. 使用亚硫酸盐,将样本DNA中没有甲基化的C全部转化为U(相当于DNA中的T)
2. 使用特殊设计的一对引物扩增样本,获得带有T7 RNA聚合酶启动子序列的扩增产物
3. 在体外转录体系中,利用T7 RNA聚合酶,将扩增产物转录为RNA片断
4. 在步骤3的体系中,利用RNase A能够特异性识别并切割RNA中U 3’端的特性,
将RNA片断切割成携带有CpG位点的小片断
5. 使用sequenom®MassArray飞行质谱分析系统检测产物。
由于同一片断中,只有CpG和CpA之间16Da的分子量差别,即质谱图中两者峰的差距。
技术优势
· 一次检测100~700bp的目标序列(一般按500bp分段)
· 可获得单个的CpG unit或CpG site的甲基化程度定量信息
· 定量数据的标准差(standard deviation)为0.05
· 由于DNaseA特异性切割U3’端,因此如果两个CpG位点之间的序列没有U(对应DNA的T),
那么这两个位点在检测时只能作为一个整体进行检测,获得的甲基化程度数值为二者的平均值
· 由于MassArray质谱平台的检测范围为1500~7000Da之间。
因此,能够检测到的片断长度在5~22nt之间。片断过长或过短的CpG位点将无法检测到。
待测序列评估案例展示
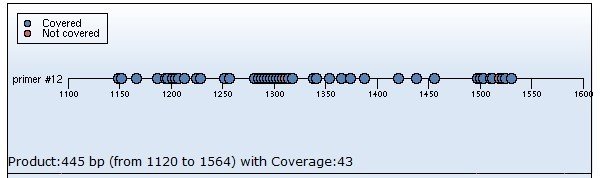
上图中序列为正向序列,圆点代表CpG位点
蓝色标记的CpG位点,可以检测到。
红色标记的CpG位点,因序列问题,无法检测到。
实验结果展示
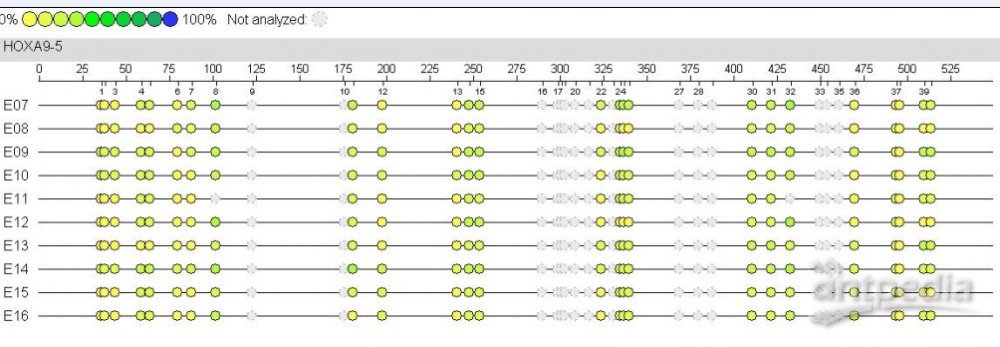
每个位点的甲基化程度另附Excel表格