研究目的
本研究的主要目的是:
在类似药物的数据集上评估ACD/Genotoxicity (AmesTest)预测模型的准确性。
将 ACD/Genotoxicity (Ames Test)的性能与市场上的几个竞争对手进行比较。
本文描述的评估基于Snyder等人[1-2]早期出版物,这些出版物侧重于评估三种流行软件(DEREK、TOPKAT和MultiCASE/MC4PC)预测药物遗传毒性的敏感性。本文的研究是这些工作的扩展,ACD/Genotoxicity (AmesTest)模型也被纳入比较范围。
实验数据
当前的研究由两部分组成,每部分都使用与先前评估之一相同的数据集:
Snyder等人[1] 原始出版物中给出的数据集包含 394 种从 2000-2002年的 PDR版本(Physician''s Desk Reference)中提取的上市药品,报告了其中 375 个分子的细菌反向诱变数据,其中 27 个(7.2%)被发现为 Ames 阳性。此外,一些化合物被排除在分析之外,因为它们产生了模棱两可的 Ames 测试结果,最终产生了 370 种药物的二元分类(阳性/阴性)数据。
在同一作者[2]最近的第二项研究中,用于分析的数据集包括 545 种上市药物,都报告了遗传毒理学数据,这些数据主要来自 1999 年至 2008 年的 PDR 版本。其中 525 种药物有定性 Ames 测试数据可用,这组诱变剂和非诱变剂的分布与之前的研究基本相同,7.1% 的测试化学品被发现会导致细菌突变。在去除产生模棱两可的 Ames 测试结果的化合物以及几种聚合物和有机金属化合物后,我们得到了最终的 516 种药物,这些药物具有指定的二元(阳性/阴性)数据。
值得注意的是,因为所考虑的化合物的性质,Snyder及其同事编制的数据集与传统上用于验证基因毒性模型的大多数数据集显着不同。几个重要方面可以概括如下:
从 PDR 中提取的化合物代表具有极低阳性率(小于 10%)的类似药物的化学空间,而计算机遗传毒性研究通常是在具有显著更高比例的显示致突变活性的化学物质的数据集上进行的。
PDR组的另一个重要特征是,其中包含的许多Ames阳性并不具有任何明显的结构特征,而这些结构特征可能是引起突变效应的原因。由于大多数基因毒性建模方法严重依赖“警示结构”的存在,因此对此类数据集实现高灵敏度的预测是一项具有挑战性的任务。
模型特点
ACD/Labs实现的Ames基因毒性预测算法是一个概率模型,即返回的预测结果是标准Ames试验中产生阳性结果的概率。该模型基于对8500多种化合物的Ames基因毒性实验数据,这些化合物以二元(阳性/阴性)形式表达。主要数据来源为知名数据库:化学致癌研究信息(CCRIS)和遗传毒理学数据库(GENE-TOX)。
该模型是使用最近开发的GALAS[3](Global, Adjusted Locally According to Similarity)建模技术建立的。这种算法引入的主要特征是模型可训练性(用户通过简单添加类似分子的实验数据来提高新化合物预测准确性的能力)和估计预测结果可靠性的内置方法,即分析的化合物是否属于模型适用范围。
可靠性评估。每一个由GALAS模型产生的预测都由一个计算的可靠性指数(RI)补充。RI值范围从0到1,提供了预测精度的定量估计。化合物的计算RI取决于以下几个方面:
与训练集化合物的相似性。如果用于推导算法的训练集中没有类似的化合物,则无法做出可靠的预测。
数据一致性。如果相似化合物的实验数据不一致,则预测质量通常较低(即对于非常相似的分子观察到相反的 Ames 测试结果)
对PDR2000-2002年数据集的评价
使用PDR2000-2002数据集的ACD/Genotoxicity (AmesTest)预测模块的准确性,结果如表1和表2所示。只有在模型适用性范围内(即RI ≥ 0.3)的化合物包括在两个表中。在表1中,阈值p=0.5用来区分positive和negative的预测结果,inconclusive代表软件没有预测结果。然而,在Snyder[1]等人的原始研究中,根据以下标准对概率TOPKAT模型的输出进行了处理:计算出的p≥0.7的化合物被认为是预测阳性; p ≤0.3预测阴性,而0.3< p < 0.7的概率被标记inconclusive,相应的化合物被排除在评估之外。为了获得公平的比较,我们对ACD/Genotoxicity (Ames Test)进行的预测也采用了相同的分类规则,得到的统计参数如表2所示。

采用文献[1]中相同的数据,将ACD/Genotoxicity (AmesTest)的预测结果精度与DEREK,TOPKAT和MCASE的结果进行了对比,结果如图1所所示。正如原始研究中所指出的,三款软件都未能检测到PDR 数据集中的大多数DNA 反应性药物(灵敏度范围为40-50%)。即使该研究获得的最高灵敏度值(51.9%)也很难被认为是可接受的,且总体趋势是这种灵敏度的增加导致了明显较差的特异性。
然而,必须指出的是,Snyder等人[1]的原始评估可以追溯到2004年,当时进行评估的三款软件均采用的旧版本的算法。随着软件的更新换代,算法也得到了显著的改进。然而在最新的出版物中,直接比较了三个主要竞争软件在同一数据集上的表现。此外,实验数据是从早期版本的PDR中收集的,可以假设在验证中使用的大多数化合物对于所有算法来说都是“已知的”。因此,并没有引入显著偏差对实验数据本身与新模型适用性进行考察。
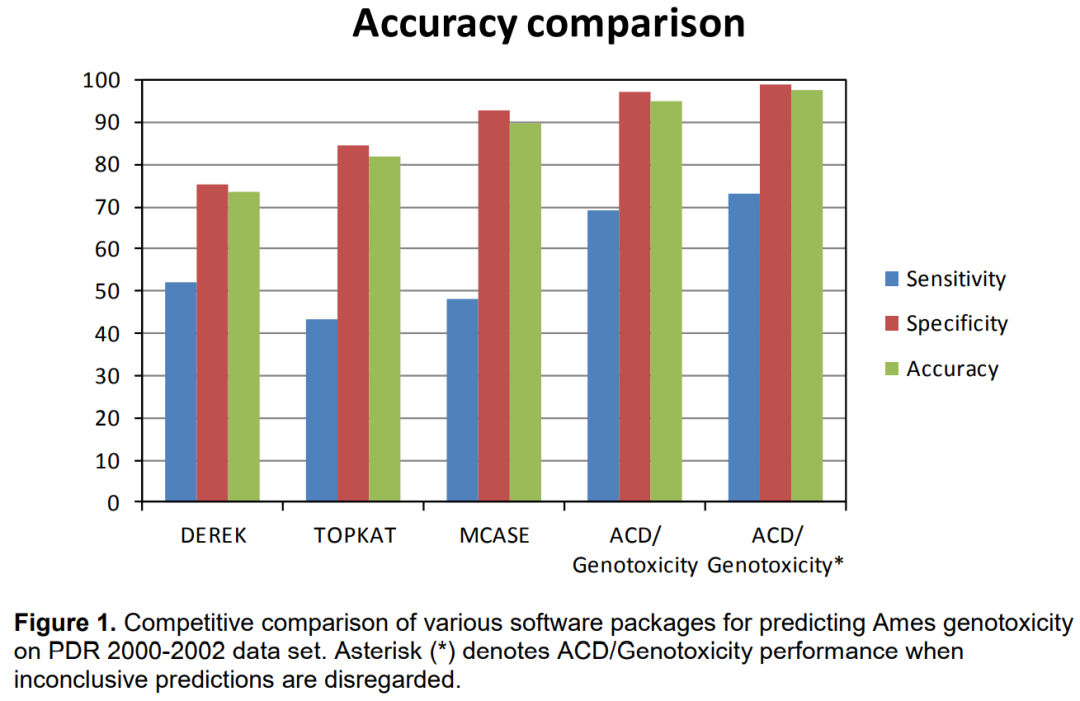
从图1可以看出,在ACD/Genotoxicity (AmesTest)中实现的模型的灵敏度较好地超过了其他软件的结果,ACD/Labs算法检测到的PDR数据集中存在超过2/3的突变原。ACD/Genotoxicity (AmesTest)灵敏度高的主要原因是能够识别非警示结构突变源,基因毒性效应的产生不是由于特定反应的亚结构的存在,而是由于整个分子的累积潜力。因此,大多数基因毒性药物(如[1]中所述)在ACD/Genotoxicity(Ames Test) 预测结果中至少有临界值,其中一些药物(氯喹、戊他汀、佐米曲坦)被确信地预测为Ames阳性。
如果在评估中不包括边界概率(p在0.3-0.7范围内)的化合物(见用*标记的条形图),情况会进一步改善ACD/Genotoxicity (AmesTest)*)。重要的是,在这些更严格的分类规则下,模型仍然适用于几乎90%的PDR数据集(见表2)。
值得注意的是,ACD/Labs模型的足够高的敏感性并不是以降低特异性为代价实现的。相反,ACD/Genotoxicity(Ames Test)对几乎所有不会引起细菌反向突变的PDR药物都进行了正确预测,总体预测准确率超过95%。
稳健模型的一个重要方面是在预测的敏感性和特异性之间保持合理的平衡。在目前对PDR基因毒性数据的验证研究中,如果考虑到模型的另一个特征——阳性准确率,就变得尤为明显。阳性准确率(或Positive Predictive Value -PPV)是一个数据集的真阳性与总阳性预测数的比率。这个参数显示了模型显示Ames阳性的化合物确实具有基因毒性的可能性有多大。
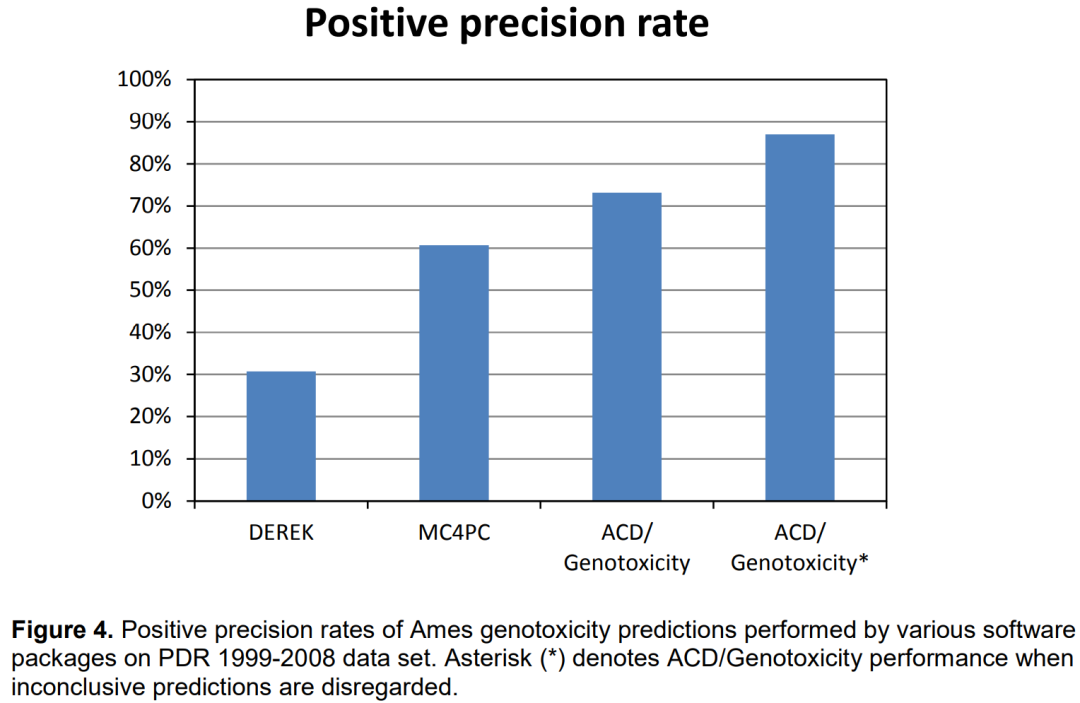
测试软件包的阳性准确率如图2所示。显然,PPV 为14.0%-16.9% 的前两个软件没有表现出药物相似物数据集中过滤潜在诱变剂的良好能力。与整个数据集(7.2%) 相比,只有MCASE 36.1% 的PPV 实现了诱变剂比例的显着(≈5x) 富集。当剔除Inconclusive结论的数据后,ACD/Genotoxicity (Ames Test)提供了更好的预测,PPV等于 66.7%。此外,当仅评估可信预测 (p ≥ 0.7) 时,PPV上升至 78.6%,这意味着在ACD/Genotoxicity (Ames Test)模型中获得高概率且具有可靠性(RI ≥ 0.3) 的5 种化合物中有4 种将通过实验确认为Ames 阳性。

对PDR1999-2008版数据集的评价
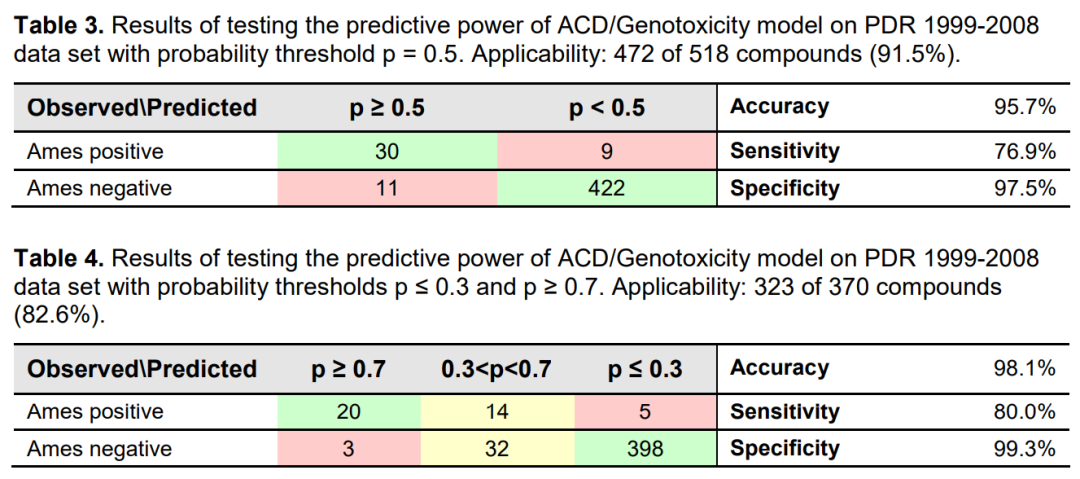
在[2]报告的扩展PDR数据集上,应用与第2.1节中描述的相同的程序来评估ACD/Genotoxicity (Ames Test)的性能。准确性检验的结果见表3(“中间区域”未定义)和表4(0.3 < p < 0.7的概率排除为不确定)。
显然,利用最近发表的文章的结果可以进行更公平的比较,其中评估中所有被考虑的软件均是最新版本。不幸的是,[2]并未评估DSTOPKAT的性能,因此只能比较ACD/Genotoxicity (Ames Test)v 2.99、DEREKv 10和MC4PC v2.0(后者对应于当前版本的MCASE软件)。图3总结了不同软件预测的精度。

获得的结果与图1中的结果非常相似。DEREK 表现出比MC4PC 更高的预测灵敏度,这被其较低的特异性所抵消。另一方面,无论用于将预测分类为阳性还是阴性的概率阈值,ACD/Genotoxicity (Ames Test)在敏感性和特异性方面始终优于其它软件。
图4显示了测试软件包的阳性准确率(阳性预测值)。与2004年的研究结果相比,DEREK和MC4PC均有显著改善。然而,在基于2009年研究的评估中,当评估所有RI≥0.3的预测时,ACD/Genotoxicity (AmesTest)产生的假阳性预测显著低于PPV为73.2%的竞争对手。当剔除inconclusive结论后,ACD/遗传毒性获得的PPV上升到87.0%,对应于9种预测为诱变物的化合物中有8种被正确识别。
值得注意的是,在2009年的基因毒预测研究中DEREK和MC4PC所证明的结果明显比2004年的好,不仅与这些软件算法的改进有关,而且与扩展PDR数据集有关。ACD/Genotoxicity (Ames Test)模型在1999-2008年PDR版本数据上所显示的所有统计参数也比使用2002年之前发布的数据所获得的各指标大约提高10%(对比图1-2和图3-4)。但是,在这两种不同数据集预测时,使用相同版本的ACD/Genotoxicity (AmesTest)。这些结果可能是由于扩展的PDR集合包含了更少的可识别性差的非警示诱变因子,因此可以更好地预测正在评估的目标对象。
值得一提的是,ACD/Genotoxicity (AmesTest)模型的训练集很好地涵盖了目前可用的毒理学文献中所代表的药物类似物化学空间,从而对大多数PDR药物进行了可靠的预测。如表3-4所示,即使在最近编制的包含新型药物代表的数据集中,超过90%的分子获得了可接受的可靠性预测(RI≥0.3),如果只考虑可确定分类为正(p≥0.7)或负(p≤0.3)的概率,仍然有超过80%的预测可适用。
上述结果清楚地表明,在预测能力方面,与主要竞争方相比,在ACD/Genotoxicity (Ames Test) 中实施的用于估计化学品的Ames遗传毒性的模型提供了更好的结果。ACD/Genotoxicity (AmesTest)的一个重要好处是能够检测非警报的诱变因子,从而对类药物数据集产生足够高的预测灵敏度,其中大多数分子(甚至那些表现出诱变活性的分子)不包含任何明确定义的危险片段。
此外,ACD/Genotoxicity模型在保持预测的高敏感性和高特异性方面表现良好。因此,ACD/Genotoxicity并没有产生大量的假阳性预测,而且在从类似药物的数据集中过滤掉潜在危险化合物方面,与竞争软件相比更有优势。
最后,值得注意的是,与竞争对手相比,ACD/Labs提供的基因毒性预测模块的好处不仅限于其更高的预测能力。ACD/Genotoxicity (AmesTest)不仅仅是一个概率模型或专家系统——它是两者的结合,使用户能够同时获得表征诱变效应可能性的数值和对其可能的原因做出基于知识的解释。
此外,用于推导模型的新型GALAS 算法引入了许多有用的特征。ACD/Genotoxicity (Ames Test)产生的每个预测都由计算出的RI 值定量评估可靠性。本研究表明,ACD软件可以对毒理学文献中目前可用的大多数化合物进行可靠的预测。然而,内部项目通常在公共资源中不具有高度特定区域化学空间的代表性。因此,第三方模型通常不适用于大多数公司的内部化合物。在ACD/Genotoxicity (AmesTest)中,这个问题由模型可训练性功能解决——通过简单输入几个类似分子的实验数据,可以立即提高对新化合物的预测准确性。最重要的是,训练过程不需要完全重建初始统计模型,这意味着可以快速轻松地扩展ACD/Genotoxicity (Ames Test)模型的适用范围以考虑用户数据。
Snyder R.D. et al. Environ Mol Mutagen. 2004;43(3):143-58.
Snyder R.D. Environ Mol Mutagen. 2009;50(6):435-50.
Sazonovas A. et al. SAR QSAR Environ Res. 2010;21(1):127-48