PART
01
和弦图的定义
和弦图是一种展示数据之间相互关系的可视化方法,和弦图主要从以下几个层面来展示关系信息:
●连接,可以直接显示不同对象之间存在关系
●连接的宽度与关系的强度成正比
●连接的颜色可以代表关系的另一种映射,如关系的类型
●扇形的大小,代表对象的度量
PART
02
和弦图的数据格式
1. 邻接矩阵
行和列分别表示连接的对象,如果对象之间存在关系,则对应行列的值将表示关系的强度。如下:
2. 邻接列表
包含三列数据,前两列的值表示连接的两个对象,第三列值为连接的强度。如下:
PART
03
和弦图简单绘制
1. 加载需要的安装包
circlize用来绘制和弦图的安装包、reshape2包是用来转换宽数据为长数据的安装包、tidyverse包是用来支持管道函数以及数据处理的包。
# 加载包
library(circlize)
library(tidyverse)
library(reshape2)
2. 生成邻接矩阵和邻接列表数据
# 设置随机数种子,确保数据可重复
set.seed(123)
#构建邻接矩阵,行为微生物,列为代谢物,值为相关性
setwd("D:/Users/Administrator/Desktop/circlize")
mat =matrix(runif(96,min=0,max=1), 8, 12)
rownames(mat) <-str_c("species",1:8)
colnames(mat) <-str_c("metabolites_test",1:12)
#构建邻接列表,第一列为微生物,第二列为代谢物,第三列为相关性
df <- melt(mat)%>%
rename(''from'' =''Var1'',
''to'' =''Var2'')
3. 简单绘制
先绘制一张简单的和弦图,绘图时我们邻接列表df。使用的核心函数是chordDiagram函数:
#使用chordDiagram函数绘制和弦图
# 使用邻接矩阵绘图
pdf("plot1.pdf",width = 12, height = 12)
chordDiagram(df)
#结束绘图,返回默认设置,否则会继续叠加图层
circos.clear()
dev.off()
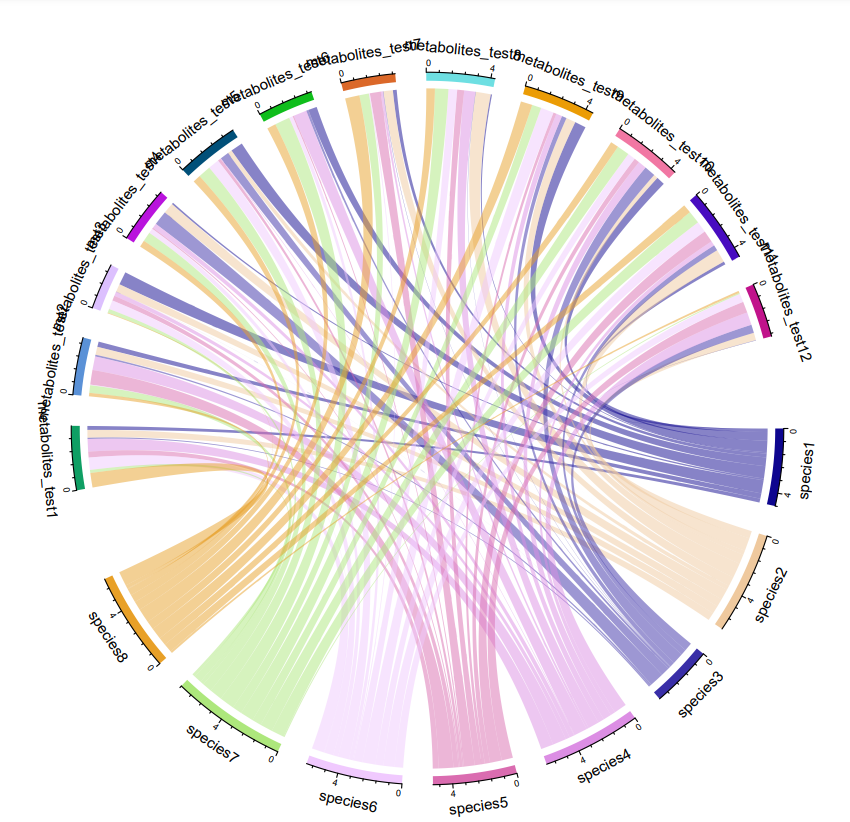
4. 绘制结果
如下图,上半截扇形为代谢物,下半截扇形为微生物,扇形的大小代表链接的度量。线条连接代表两者有相关联系,其宽度与关系的强度成正比。
PART
04
和弦图的细节调整
目前为止,图片基本完成。但是经常会对扇形间距、扇形顺序,扇形颜色、链接颜色、字体方向、字体大小做一定的调整。代码和注释如下:
pdf("plot2.pdf",width = 18, height = 18)
# 调整外围sectors的排列顺序
orderlist <-c(rownames(mat), colnames(mat))
#设置不同sector之间gap的间隔大小
circos.par(gap.after= c(rep(5, nrow(mat)-1), 15, rep(5, ncol(mat)-1), 15))
# 设置扇形颜色,要和对应的名称对上
colors <-c("#C6CF85", "#717862", "#943A74","#7267AC", "#040000",
"#DA3918", "#172F2E", "#80B862","#635368", "#DAEDDF")
colors <-colors[1:nrow(mat)]
col_list1 <-c(colors, rep("grey50", ncol(mat))) %>%`names<-`(c(rownames(mat), colnames(mat)))
# 设置link颜色,link颜色与列所代表的扇形颜色一致,因此使用column.col参数设置
col_list2 <-colors %>% `names<-`(rownames(mat))
chordDiagram(df,
order =orderlist, #设置扇区顺序
grid.col = col_list1, #设置扇区颜色
column.col = col_list2, #设置link颜色
transparency = 0.3, #设置不透明参数
annotationTrack = c("grid", "axis"), #设置外围类型,可从c("name","grid", "axis")中指定任意值
preAllocateTracks = list( # 提前对轨道的占比进行定义
list(track.height = 0.05),
list(track.height = 0.005)
))
# 注释区呈放射状,函数固定写法
circos.track(track.index= 1, # 轨道索引
panel.fun =function(x, y) {
circos.text(CELL_META$xcenter, # x轴上的数据点
CELL_META$ylim[1], # y轴上的数据点
CELL_META$sector.index, # 轨道索引
facing= "clockwise", # 文本面向c("inside","outside", "reverse.clockwise","clockwise","downward", "bending","bending.inside", "bending.outside")
niceFacing = TRUE, # 文本的正面是否应该调整以适合人的眼睛
cex =1, # 字体大小
adj =c(0, 0.5)) # 文本的偏移量
}, bg.border = NA)#这个不能缺
circos.clear()
dev.off()
调整完图片如下:

PART
05
和弦图的教程链接
https://github.com/jokergoo/circlize直播预告

99%的代谢组学研究者都在阅读下文(精彩合集,欢迎收藏):
●激情世界杯,追逐科研梦 | 转录组+代谢组数据挖掘与R语言培训班
●脂肪酸专题:一文了解脂肪酸的分类、合成途径及相关疾病
●蛋白专题合集
●Biomarker专题合集
●空间代谢组合集
●肠道菌群&微生物专题
●生信小工具专题

客服微信:metware888
咨询电话:027-62433042
邮箱:support@metware.cn
网址:www.metware.cn
我就知道你“在看”