RNA 测序如何做好样本质控,单看 RIN 值就足够了吗?为什么有的时候 RIN 值挺高的,但有些位点却怎么也测不到?研究小 RNA 的差异表达时是否可以参考 RIN 值?
针对这些 RNA 测序应用中经常遇到且困扰大家的问题,经验丰富的华大科技在 18 年夏天的直播课程中结合文献与华大的经验给大家做了精彩的分享。经由华大许可,我们将直播课程中的精华部分组织成了文字内容与大家分享。(讲师:宗亮,华大科技技术优化高级工程师,主要负责 RNA 测序技术的工艺优化及 Troubleshooting、自主测序平台的技术转移和技术支持。著有 SCI 文献一篇,发明专利四项。)
RNA 完整性常用参数 —— RIN 值
RIN 值是安捷伦公司开发的以数字化形式表示 RNA 完整性的参数。
RIN 值相比核糖体 18S:28S 的结果可信度更高
RIN 值反映总 RNA 的完整性,与样本浓度无关
RIN 是基于以人、小鼠、大鼠为代表的哺乳动物开发的

图 1. RIN 值比 18S:28S 更可靠,不受样本浓度的影响。图中原始样本的核糖体 CV 为 5.1%,RIN 值为 8;将样本梯度稀释后,核糖体 CV 变为 22%,而 RIN 仍为 8,说明 RIN 值的稳定性明显优于核糖体比值。
RNA 的质量受多因素的影响
Ambion 的操作手册中讲到,RNA 的完整性对 GLOBIN 清除步骤以及下游应用的成功至关重要,即使是中等水平的 RNA 降解也会导致捕获试剂在清除球蛋白 mRNA 时的不充分
血液样本的保存、RNA 纯化的方法,以及残留 RNA 酶的活性都会影响 RNA 的质量
Illumina 的产品手册中要求 RIN 值要大于8,华大根据交付样本的大数据分析,建议交付的样本的 RIN 值大于7(关于 RIN 值的标准,用户可以根据现实研究经验进行设定)
RIN 值可结合 RT-PCR 来对用于表达谱的 RNA 进行质量评估
挪威公众健康中心为了研究如何将全国各地的样本安全送到样本库而免受人为因素的干预,除了采用合适的采血管、最佳 / 次优的采血体积,同时还分析了来自全国各地样本的表达谱差异1。
通过 RIN 分析总 RNA 的完整性
对 6 个有代表性的与癌症发生相关的基因进行 RT-PCR 分析其转录本的变化
研究的结果是 RNA 质量未受运输过程的影响;关键基因的转录本的表达模式没有发生显著变化
这种通过 RIN 值检测,结合 RT-PCR 检测关键基因转录本的稳定性可以更全面的评估 RNA 的质量,特别是样本是否适用于下游表达谱研究。
华大专家友情提示:当用 RT-PCR 对 NGS 基因表达变化的结果进行验证时,需要注意三点:
1. 如果使用 Oligo dT 引物做反转录,要同时注意样本的 RIN 值,以及设计扩增子时注意其与转录本 3’端的距离
2. 用于 RT-PCR 验证的样本尽量与 NGS 检测用的样本来自同一样本管,以最大限度保证 RIN 值一致,并使用与转录组文库构建时相同的逆转录方法
3. 尽可能验证每对引物的特异性与扩增效率,包括文献已经报导的引物设计
融合基因检测时除了 RIN 值,还要考虑融合位点与 3’端的距离
影片《我不是药神》中慢粒白血病的靶向药格列卫靶向融合基因 BCR-ABL。有研究表明在 BCR-ABL 阳性患者中有不到 80% 患者同时也存在 ABL-BCR 融合。在梅奥诊所的研究中,当样本 RIN 值为 10 时,测序可以同时检测到两种融合产物,而当 RIN 值为 7 以下时就检测不到 BCR-ABL 了2。
从梅奥诊所的这项研究结果可以推断出两个融合位点距离 3’端是有差异的,而事实上的确如此,BCR-ABL 距离 3’端 5kb,ABL-BCR 融合距离 3’端只有 1.5kb(图 2, b)。将整个测序数据平铺在转录本上,横坐标是融合位点与 3’末端的距离,纵坐标是覆盖的 reads 深度。结果显示:
只有当 RIN 值为 10 时,几乎可以实现对距离 3’端 0-5kb 区域的均匀覆盖(图 2, c)
一旦 RIN 值小于 10,就很难覆盖到距离 3’端大于 2kb 的区域了(图 2, c)
图 2, d 中,当 RIN 为 3 时,即使距离 3’端 1.5kb 的 ABL-BCR 也不能被很好的覆盖
梅奥在研究中同时指出:
COSMIC(人癌症变异)数据库中,目前已知的融合基因产物的融合位点距离 3’端的中位数为 2.7kb,大约有 20% 融合位点距 3’端大于 5kb;5% 距 3’端大于 7kb
基于梅奥的研究结论:RIN 值小于 10 时,难以测到距离大于 2kb 的融合位点。也就是说当 RIN 值小于 10 时,至少有超过 20% 的融合基因的产物无论测多少深度都是测不到的
融合位点的成功检测基于两个因素:RIN 值、融合位点与转录本 3’端的距离
梅奥研究的提示:在做转录本研究时(特指用 Poly A 钓取法研究编码 RNA 时),RIN 值不仅能提示 RNA 的质量,同时能对测序数据做出预期。
图 2. 从这张 PPT 里可以看出,距离转录本 3’端更近的 ABL-BCR 更容易被检测到;只有当 RIN 值为 10 时,可以对距离 3’端 1-5kb 区域实现均匀覆盖;当 RIN 值小于 10,就很难覆盖到距离 3’端大于 2kb 的区域。
在以 PolyA 钓取为主要技术手段研究编码 RNA 时,RIN 只能做为评估基本样本完整性的方法,而不能预测表达谱数据的可信度
在两组独立的实验中,实验 1 将 UHR(人标准参照)处理成不同 RIN 值的梯度,针对不同基因位点的测序结果做散点检测,以基因位点距转录本 3’端的距离和检测灵敏度(有效 reads 数)分别为横坐标和纵坐标3。结果发现 RIN 值相同(7.5)的两个不同样本在做散点图时,一个的曲线更拟合于 RIN 值为 8.6 的 UHR,另一个的更拟合于 RIN 值为 5.9 的 UHR 样本(图 3)。实验 2 对健康志愿者的外周血分离单核细胞,在室温下静置不同时间(形成不同降解程度),纯化后进行以 PolyA 为技术手段的转录组测序4。对结果进行聚类分析,维度一为处理方案,即静置时间,维度二为样本的 RIN 值。在聚类结果中发现,聚类依据更多的与样本处理方案相关,而非 RIN 的差异(图 4)。
从这两份研究中得到的结论是:
1. RIN 值可以做为基本样本完整性的方法,但不能预测某一个特定基因的完整性,这种情况下测序数据是样本完整性的较为客观的反映
2. 在大多数情况下 RIN 值不能预测表达谱数据的可信程度
3. 表达谱数据的改变与取样方式、保存方法,以及提取方法之间有着显著的关系
4. 研究人员最好能够配合多种方法学的验证,以得出更准确的实验结论

图 3. 两个 RIN 值相同的不同样本,它们的表达谱数据与标准对照的聚类结果并不相同。

图 4. 转录组测序的聚类结果与样本的 RIN 值并没有呈现出显著的相关性。
RIN 值低的样本也可能用于小 RNA 测序
加拿大的研究者在严格控制的实验环境下(没有核酸酶污染),以不同起始量的总 RNA 构建测序文库;并对总 RNA 进行处理,获得 RIN 值从 9-2 的样本5。测序结果表明:
1. RNA 测序鉴定到的小 RNA 的数目与建库时所用的起始总 RNA 量没有显著关系
2. RNA 测序数据的可利用程度(用于鉴定小 RNA)与 RIN 值没有显著关系
3. 以全血为实验材料时,小 RNA 具有极高的稳定性,样本 RIN 值低也可以做小 RNA 测序,并且小 RNA 的定量结果是很可靠的
另一项研究发表在 Nature Biotechnology 上,研究者在全美选择了 9 个技术过硬的实验室,用 4 家主流的 RNA-Seq 试剂盒(实验方案),对体液的小 RNA-Seq 结果展开对比6。图 5 中,三百多条合成序列(浓度设定已知)分为 A 组和 B 组。结果显示:
1. 聚类的结果只与建库的方法有关,如果建库方法不同则同一实验室同一样本的结果也不一致
2. 当只关心小 RNA 的差异表达时,最右侧的结果显示不同方法与实验室之间具有很好的可比性
3. 当以血清样本替换合成样本时,结果一致。说明以血清为代表的液体活检,当只关注小 RNA 的差异表达时,结果是有很好的可重复性和可再现性的。
上述两组实验的结论可汇总为:
小 RNA 具有极高的稳定性,样本 RIN 值低也可以做小 RNA 测序,且定量结果可靠
小 RNA 的检测结果与建库方法有关
当只关心小 RNA 的差异表达时,即使实验方法、批次,甚至由不同公司开展检测,其数据的可信度和可比性仍然很高

图 5. 结果显示,相同的实验方法之间更强的相关性;与之相对的是,当分析 A 与 B 的相对值时,使用相同实验方法的不同实验室显示出非常高的相关性,并且即使在不同方法间也显示出高度的相关性。这说明相对定量对实验方法的差异有更好的包容性。
FFPE 样本的质量评估
FFPE 样本最具挑战性。Illumina 推荐使用 DV200 来评估 RNA 是否可用于转录组测序,他们认为 DV200 大于 30%,即总 RNA 中大于 200nt 的组分占比大于 30% 时是可以用于转录组测序的
针对不同完整性的 FFPE 样本,我们可以通过选择不同的实验方案来实现对样本内核酸信息的最大限度挖掘
诺华制药生物医学研究所针对不同样本用量、不同降解程度,以及不同 RNA 实验方法设计了一个宏大实验方案,用于评价最优的建库方案7。基本从五个维度进行了全面的评价:
1. 能否产生高质量的测序数据;
2. 对于已知转录本是否能够实现全长的覆盖;
3. 能否检出非编码的 RNA 序列(这对于医学研究特别重要);
4. 对于已知的基因,其定量的结果是否准确,例如与 QPCR 的方法或其它的方法学的结果相比较;
5. 当使用不同的样本量、降解程度时,其定量结果是否是可再现的(图 6)。
这篇文章的结论是:在应对不同起始量、不同 RNA 完整程度的样本时,不同的建库方案都有各自的优缺点;但针对不同完整性的样本,我们可以通过选择不同的实验方案来实现对样本内核酸信息的最大限度挖掘。文章提到,当使用 TruSeq 的建库方法,即使样本只有 5ng 时,其测序结果也不错;而对于严重降解的样本,文章认为 Access 的方法可能是更好的选择。
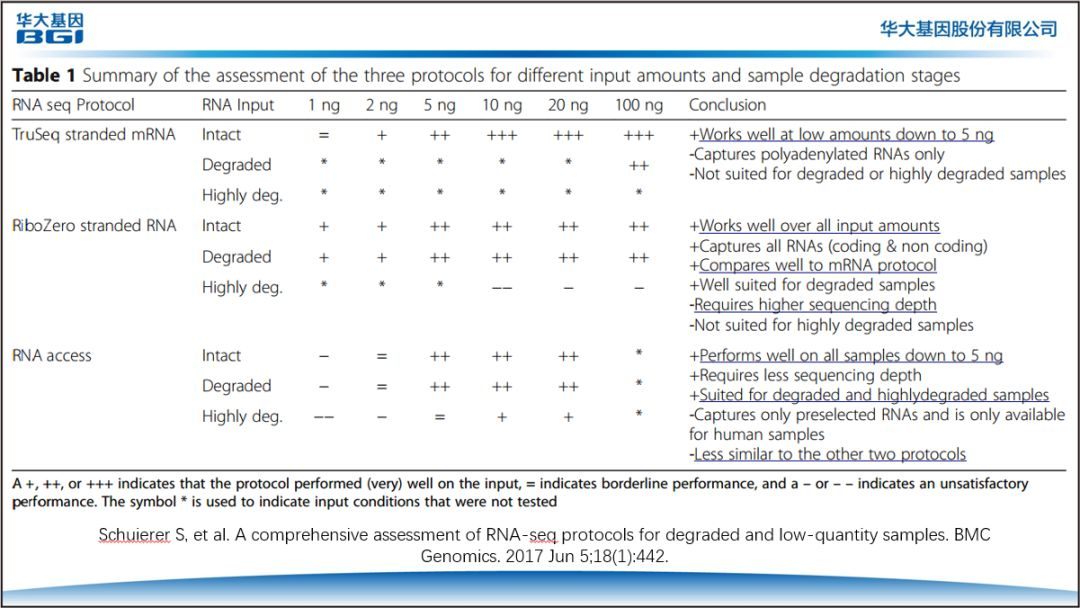
图 6. 对不同样本用量和降解程度的样本,以三种不同实验方案进行 RNA 测序,得出的比较结果与建议。
篇幅所限,我们无法百分百展示华大本次课程的全部内容。在本文的结尾,我们结合了华大专家的建议与安捷伦对 RNA 质控的经验与理解,与大家分享 RNA 质控的要点:
1. RIN 值是评价 RNA 完整性的最为广泛应用的工具
2. 在对转录本进行研究时,转录本的完整程度并不能与 RIN 值建立起直接关系,特别是针对某一条或某一些特定转录本时,其不相关性可能更为突出
3. 在做表达谱研究时,可以将 RT-PCR 与 RIN 值结合在一起评估 RNA 的质量
4. 以 Poly A 钓取方法检测基因融合时,除了 RIN 值,还要考虑融合位点与 3’端的距离
5. 小 RNA 具有极高的稳定性,样本 RIN 值低也可以做小 RNA 测序,但检测结果与建库方法有很大相关性
6. 实验设计时可以根据样本的类型、起始样本量、完整程度等对生信方案,建库流程进行调整以获得最优的测序结果
参考文献:
1. Duale N, et al. Human blood RNA stabilization in samples collected and transported for a large biobank. BMC research notes. 2012;5:510.
2. Jaime I, et al. The impact of RNA degradation on fusion detection by RNA-seq. BMC Genomics. 2016; 17: 814.
3. Winters JL, et al. Development and Verification of an RNA Sequencing (RNA-Seq) Assay for the Detection of Gene Fusions in Tumors. J Mol Diagn. 2018 Jul;20(4):495-511.
4. Gallego Romero I, et al. RNA-seq: Impact of RNA degradation on transcript quantification. BMC Biol. 2014 May 30;12:42.
5. Lopez JP, et al. Biomarker discovery: quantification of microRNAs and other small non-coding RNAs using next generation sequencing. BMC Med Genomics. 2015 Jul 1;8:35.
6. Giraldez MD, et al. Comprehensive multi-center assessment of small RNA-seq methods for quantitative miRNA profiling. Nat Biotechnol. 2018 Sep;36(8):746-757.
7. Schuierer S, et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 2017 Jun 5;18(1):442.