
如何查找参考基因组及选择最优参考基因组
前言
随着高通量测序技术的不断发展,越来越多物种的基因组得到组装和注释,同一物种的基因组版本也越来越多。许多老师做完转录组测序后,经常不知道自己的物种是否有参考基因组以及应该选择哪版参考基因组,下面介绍几个常见参考基因组查找数据库以及下载方法。
常见参考基因组下载网站
1. Ensembl(最优选)
Ensembl 是由 European Bioinformatics Institute(EBI)与Wellcome Trust Sanger Institute(WTSI)共同合作开发的数据库项目。涵盖大量物种的参考基因组信息,并且数据更新及时,是参考基因组下载的好选择。
动物参考基因组:http://asia.ensembl.org/index.html
植物参考基因组:http://plants.ensembl.org/index.html
其他真菌细菌等参考基因组:http://ensemblgenomes.org/
2. NCBI (最全面)
NCBI是National Center for Biotechnology Information的缩写,指美国国立生物技术信息中心。NCBI的数据非常全面丰富,在参考基因组信息展示上同样表现出色。
地址:https://www.ncbi.nlm.nih.gov/genome/
3. UCSC
UCSC Genome Browser是由University of California Santa Cruz (UCSC) 创立和维护的,主要收录一些模式动物的数据库,尤其是人和鼠参考基因组较常用。关于人的基因组注释信息非常全面。
地址:http://genome.ucsc.edu/cgi-bin/hgGateway
4. phytozome(JGI)
主要收录绿色植物基因组的数据库,主要用于植物比较基因组学分析,收录的植物基因组及注释信息很全面,也是一个不错的植物基因组下载数据库。
地址:https://phytozome.jgi.doe.gov/pz/portal.html
5. plaBiPD
plaBiPD是一个专门进行植物基因组检索的网站,在该数据库中可以根据发表时间、进化关系进行检索,可以查看每个植物基因组发布的时间、基因组大小、发表期刊信息。
地址:https://www.plabipd.de/index.ep
参考基因组查找实操
对于大部分物种来说,Ensembl数据库具有注释信息详细,操作简单的优点,所以可以首选Ensembl数据库进行参考基因组选择,其次选择NCBI。下面我们以Ensembl 和NCBI 两个网站为例,示例演示一下如何查找拟南芥(Arabidopsis thaliana)的参考基因组。
Ensembl数据库参考基因组查找
1. 输入地址 http://plants.ensembl.org/index.html 进入植物参考基因组查找页面(左上角点击箭头也可以切换到其他动物,细菌,真菌等参考基因组页面),右下区域Favourite genomes 可以自定义展示参考基因组;也可以在左边下拉框选择参考基因组物种,或者点击View full list of all species 查看网站收录的所有植物参考基因组。

2. 可以根据左侧物种拉丁名选择老师研究的物种基因组,也可以在右侧方框内输入物种拉丁名进行检索。

3. 点击拟南芥物种拉丁名,进入拟南芥的基因组信息展示页面,可以查看基因组的组装注释信息,点击Download DNA sequence (FASTA) 可进入到参考基因组序列信息链接; 点击GFF3可以进入参考基因组注释信息链接

4. 复制基因组序列信息链接和基因组注释链接地址,填写到我们提供的信息采集单即可。
4.1 地址栏全选,复制基因组序列地址链接。

4.2 地址栏全选,复制基因组注释地址链接。

4.3 将参考基因组序列和注释信息地址填写到我们提供的信息采集单地址栏。

NCBI数据库参考基因组查找
1. 输入地址 https://www.ncbi.nlm.nih.gov/genome/ 进入NCBI参考基因组查找页面,输入拉丁名Arabidopsis thaliana 检索拟南芥参考基因组信息。

2. 在检索页面可以直接点击genome下载拟南芥基因组序列、点击GFF下载基因组注释文件也可以点击 RefSeq 进入到拟南芥基因组ftp 地址,查看所有基因组序列和注释信息,基因和蛋白序列等信息。

3. 在gemome或者GFF处鼠标右击,在打开的页面复制链接地址,然后将拟南芥基因组链接填写到我们提供的信息采集单相应位置。
3.1 在gemome或者GFF处鼠标右击,选择复制链接地址(对于存在多个参考基因组版本的物种来说,genome 和GFF处链接提供的是由NCBI工作人员选择并维护的RefSeq版本的参考基因组)。

3.2 将拟南芥基因组序列和注释文件地址填写到信息采集单相应位置。

参考基因组评估
在掌握了参考基因组查找和下载的方法后,通过NCBI数据库下载基因组时,有时候我们会遇到一个物种有多个版本的参考基因组的情况,这种情况下该如何选择了?下面我们一起来看一下如何选择合适的参考基因组。
1.RefSeq or GenBank
NCBI数据库GenBank版本的基因组和RefSeq版本的基因组区别:
GenBank genome 一般以GCA_xxxx 为起始 ,注释信息可有可无,一般由提交参考基因组版本的人员进行维护,geneID格式不统一;
RefSeq 的基因组版本一般以GCF_xxxx为起始,是由NCBI工作人员选取的参考基因组,手动注释修正,高质量,有注释,NCBI负责维护,所以在进行参考基因组版本的选择是可以优先推荐RefSeq版本的参考基因组。
下图更详细的说明了NCBI数据库GenBank版本基因组和RefSeq版本基因组的区别。

2.通过NCBI Datasets 选择合适参考基因组
除了选择RefSeq版本的参考基因组之外,还有哪些筛选标准帮我们找到合适的参考基因组呢?下面介绍如何通过NCBI Datasets 的筛选功能来选择合适的参考基因组。
1. 如何查看NCBI数据库物种不同版本的基因组?
通过NCBI找到参考基因组之后,如果存在多个基因组版本,点击NCBI Datasets,会进入到基因组不同版本界面。

2. 如何选择和自己研究品种相近的参考基因组
对于某些物种,NCBI数据库中也可能会存在多个不同品种的参考基因组,那么如何选择和自己研究品种最相近的参考基因组呢?输入物种拉丁名,根据第三列的品种信息可以查看和自己研究品种相近的基因组版本。

3. 还可以通过Filters 栏中 STATUS ,ASSEMBLY LEVEL , TEXT FILTER 和YEAR RELEASED四个选项通过是否有NCBI官方的注释信息,基因组组装水平,发布时间等信息来筛选参考基因组。

1)点击 reference genomes 前的按钮,可以看到只有一条信息,这个也是NCBI负责维护的RefSeq版本的(RefSeq: GCF_000001735.4) 参考基因组。(注:并不是所有的物种都有该版本的基因组注释信息)
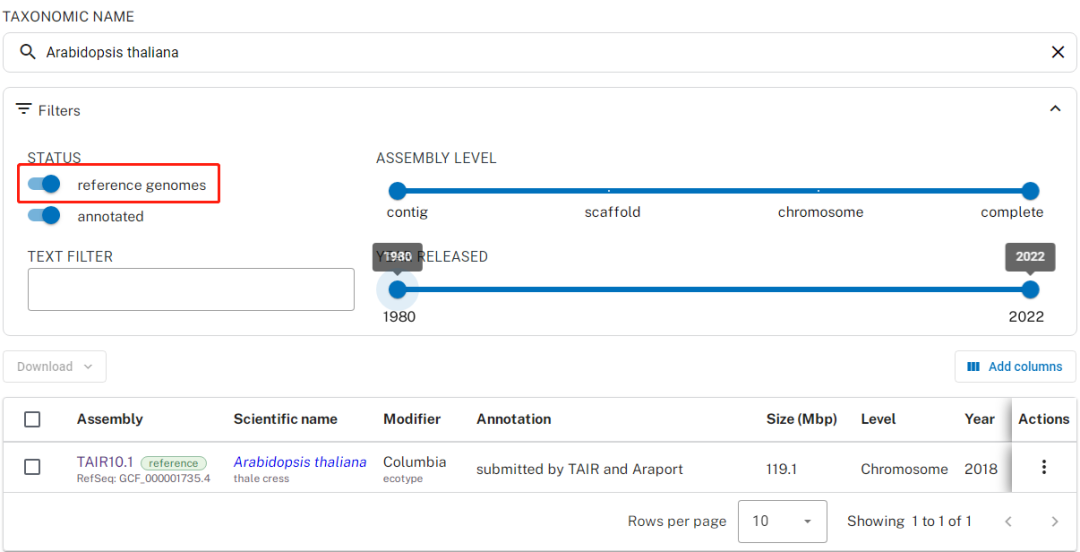
2)点击 annotated 前的按钮,选择NCBI Datasets 中有GFF注释的基因组,ASSEMBLY LEVEL 选择染色体水平,YEAR RELEASED 选择2013-2022年, 可以看到有四条信息。

3)点击第一列TAIR10.1 、Ler Assembly 或AT9943.Cdm-0.scaffold 的链接可以查看任意版本基因组组装注释详细信息,可以通过Assembly statistics、Sample details 和Annotation details 进一步选择基因组。
a. Assembly statistics 可以查看Scaffold N50 、Contig N50 等信息,一般而言N50值越大说明组装质量越高。

b. Annotation details 可以查看基因数量,一般而言基因越多表示注释越完整。

3.通过GFF/GTF文件评估基因组注释完整性
GFF全称Generic Feature Format, 描述了基因组上各种特征的区间信息,包括染色体,基因,转录本等。GFF文件本质上是一个\\t分隔的,共9列的纯文本文件。GFF3 格式说明见 http://gmod.org/wiki/GFF3 。下载GFF/GTF文件后,使用Notepad++等文本文件打开,仔细检查GFF文件,若显示有exon和CDS信息,则基因组注释相对完全,可用于分析。如果既没有exon信息,也没有CDS信息,则基因组注释信息缺失,需要等注释信息完善后才可以用于有参转录组分析的参考基因组。

4.通过Hisat评估序列一致性
通过Hisat2 等比对软件将测序reads 比对到下载的参考基因组序列,统计比对率,比对率越高说明测序reads和下载的参考基因组序列一致性越高。在参考基因组注释信息都比较完善的情况下,优先推荐比对率高的基因组版本。
结语
综上所述,在有参考基因组GFF注释文件的前提下,如果老师研究的物种存在多个不同的品种,我们可以优先选择和自己研究的品种更接近的参考基因组版本。如果只有一个品种,我们可以直接选择由NCBI官方维护的RefSeq版本的基因组,或者结合基因组组装水平,发布时间,注释完整性来进行选择。

精彩合集,欢迎收藏
迈维云平台系列课操作教程,公号回复“迈维云”即可

客服微信:metware888
咨询电话:027-62433042
邮箱:support@metware.cn
网址:www.metware.cn
我就知道你“在看”
