简化基因组测序利用限制性内切酶获取基因组上与酶切位点关联的序列片段,进行高通量测序,获得海量标签序列来代表研究物种的全基因组信息。常见的简化基因组技术主要有RAD(Restriction site Associated DNA)、GBS(Genotyping By Sequencing)等。主要目的是开发分子标记,进而进行种质资源鉴定、群体进化、GWAS、图谱构建、BSA定位等,为功能基因的挖掘及标记辅助育种提供理论基础。
应用领域:
遗传图谱构建及QTL定位
群体遗传学分析
局部组装及新分子标记开发
产品优势:
相对于全基因组重测序,成本相对低,研究所需数据量低,极大简化了基因组
对于大量样本、基因组比较大、高杂合或研究相对落后的物种具有极高的性价比
检测范围广,可适用于各类育种群体,有无参考基因组均可进行研究
无参考基因组:不受已知基因组序列的限制,可大规模筛查SNP位点,降低基因组 的复杂度,测序成本降低,适合在群体水平进行研究
有参考基因组:数据分析简便,且能够有效开发新的SNP位点
技术路线:
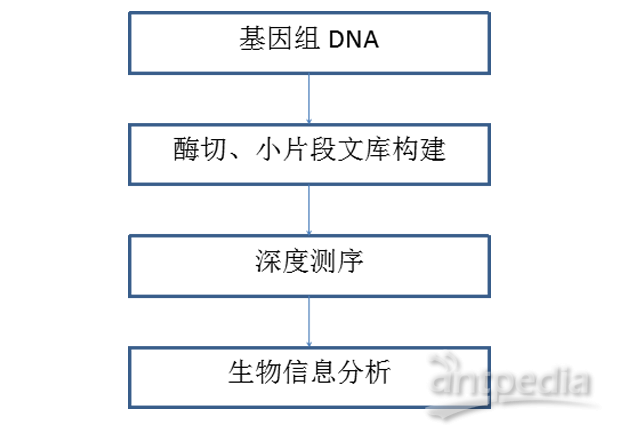
1. 标准信息分析
a) 测序数据基本分析
b)个体RAD/GBS tag聚类及统计
c)群体RAD/GBS tag 聚类
d) SNP及Indel检测
2. 高级信息分析
a)饱和度分析
b) Contigs组装(利用Paire-end read 的read2)
c)利用Contigs检测SNP,Indel及SSR,并进行PCR引物设计
d)群体变异频率统计(为后续大群体验证挑选标记提供参考)
e)群体进化树分析
f)群体结构分析(利用Structure软件分析群体间的可能基因交流)
g)群体主成分分析(分析群体聚类关系)
h)遗传图谱构建及QTL分析
i)遗传图谱整合(需提供同一作图群体原图谱数据)
研究路线:
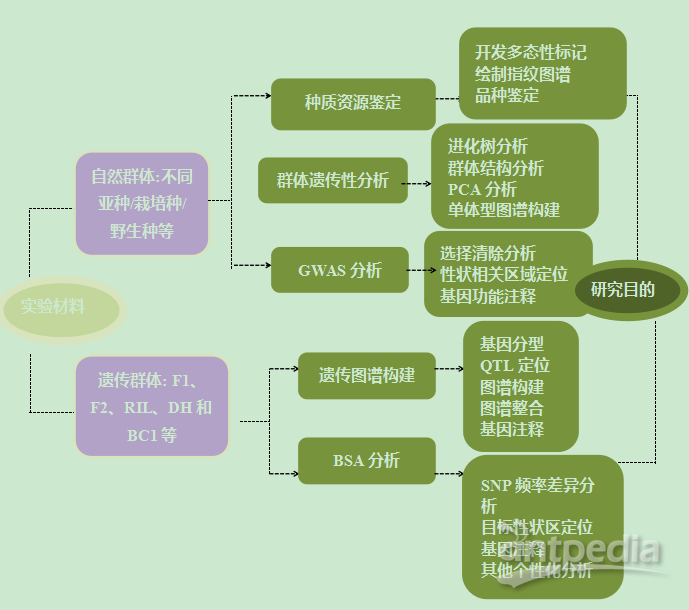
样品要求:
1)样品类型:无降解且无RNA污染的DNA样品;
2)样品需求量:≥2μg;
3)样品浓度:≥25 ng/μl;
4)样品纯度:OD260/280=1.8~2.0;
5)推荐样本大小:遗传图谱构建>100,群体进化>30。
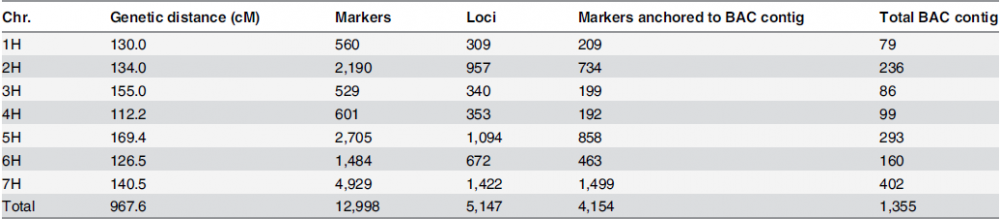
1、RAD-Seq构建大麦高密度遗传图谱
本研究对两个品系的大麦Baudin、AC Metcalfe及其杂交产生的94株RILs后代进行RAD-Seq测序,并进行高密度遗传图谱构建:12,998 个高质量的SNP,总长为967.6cM(遗传距离)的高密度遗传图谱,用于后续QTL定位、候选基因的筛选以及辅助全基因组序列的组装等。该研究思路也有助于其他物种研究快速地建立高密度遗传图谱。
各染色体SNP标记统计
2、RAD-seq研究三刺鱼群体进化与地理分布
本研究运用混池RAD-seq,研究了波罗的海10个不同三刺鱼种群的全基因组变异和分化模式,且种群之间差异很大,这可能与温度及盐分梯度密切相关,且发现了与环境适应的候选基因,很可能是与生活的咸水和淡水环境有关。总的来说研究结果为不同种群基因型差异提供了有力证据:波罗的海形成过程中基因流随着环境变化逐渐产生环境适应性表现。
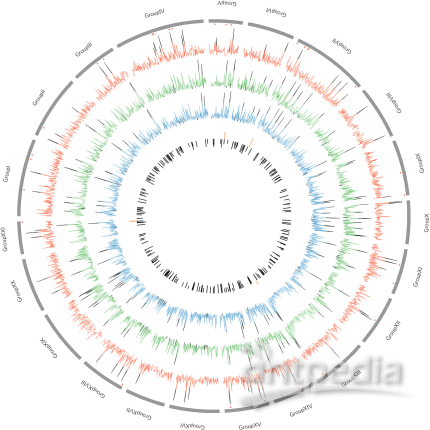
10个不同三刺鱼种群的全基因组变异和分化模式
参考文献:
1. Construction of High-Density Genetic Map in Barley through Restriction-Site Associated DNA Sequencing (PLOS ONE, 2015, IF =3.234)
2. Population genomic evidence for adaptive differentiation in Baltic Sea three-spinedsticklebacks (BMC Biology, 2015, IF = 7.984)