现代系统生物学有两种革新技术:基因组学和单细胞生物学,前者具备同时监测生物体中所有基因和蛋白质的能力,后者则可以在自然微环境中跟踪单细胞的一些特定基因。
两种技术都很强大,但具有互补的局限性:基因组学平均了一个细胞群的异质性和空间复杂性,而单细胞技术一次只能探测几个基因。因此,将基因组学与单细胞结合的生物学方法是下一阶段的主要挑战。生物和生物工程教授Long Cai致力于开发合成基因组学与单细胞的革新方法。
通过序列杂交和条形码进行单细胞原位分析
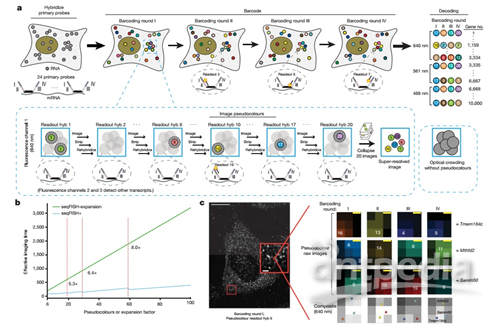
过去几年,Cai教授实验室采用FISH的原位“测序”来分析单细胞的基因表达。为了检测每一个mRNA,他们的序贯FISH(seqFISH)策略在概念上类似于用FISH探针对单个细胞中的转录序列进行测序,用FISH探针进行序列杂交,对mRNA进行编码。在一轮杂交过程中,每个转录物都被一组标有单一荧光团的FISH探针作为目标,然后对样品进行成像,利用酶消化去除FISH探针。在随后的一轮采用相同的FISH探针杂交,但使用不同的染料标记。由于转录物是固定在细胞中的,多轮杂交过程下来,对应单个mRNAs的荧光点仍保持在原位,并且可以对齐以读取颜色序列。因此,每种mRNA都被分配到了一个独特的条形码,通过计算相应条形码的数量就可以确定给定单元中每个转录本的数量。
seqFISH的可用条形码数量理论上可以覆盖整个转录组(6轮杂交),但需要超分辨率显微镜来解析细胞中的所有转录情况。Cai课题组最近发表在《Nature》杂志上的文章介绍了改进版seqFISH+,其仅需利用序列杂交和普通共聚焦显微镜即可获得单个细胞内不少于10,000个基因的多路复用和超高分辨率成像。
seqFISH+的关键在于扩展的条形码库。seqFISH平台使用四种或五种颜色染料,升级版技术则拥有更庞大的“伪颜色”调色板(图1)。通过使用60个伪颜色通道,研究人员有效地将mRNA分子稀释成60个单独的图像,并在重组图像以重建超分辨率图像之前使每个mRNA点定位于衍射极限以下。
60种伪颜色被分为3个荧光通道(Alexa Fluor 488、Cy3b和 Alexa Fluor 647),并且只在每个通道内生成条形码,以避免通道间色差。每个通道可包含8000个被打上条码的基因,当用伪颜色成像四次时(其中一轮用于纠错),理论上可获得24,000个基因的信息,与seqFISH相比,成像时间缩短了8倍。
最初,研究团队在纯净的NIH/3T3成纤维细胞中检测seqFISH+的实际效率。随机选取10,000个基因(排除高丰度的看家基因),结果每个细胞大约获得了35,492±12,222个转录本,其中60个基因的单分子荧光原位杂交(smFISH)结果显示,其灵敏度比单细胞转录组测序(scRNA-seq)更高。
研究人员进一步使用相同的10,000个基因探针观察了小鼠脑室下区(SVZ)皮层和嗅球独立组织切片,总共收集了2,963个细胞中10,000个基因的转录图谱(图2)。
为了节省时间,本文只观察了一个光学平面(0.75μm厚),如果从3D角度观察,每个细胞的可检出转录本将是文中的5-8倍。在无监督聚类分析处理下,seqFISH+细胞簇清晰地呈现出层状结构,与scRNA-seq数据集强烈相关。
在seqFISH+的帮助下,人们有能力以细胞类型为单位直接探索脑内上万mRNAs的亚细胞定位模式。在本文中,科学家们定量了SVZ和嗅球内不同细胞类型的空间组织结构。此外,还分析了富含相邻细胞信息的配体-受体对,在过去,研究人员无法从分离细胞中获得这些信息。事实上,这些潜在的细胞-细胞相互作用基于mRNA而非蛋白质。令人意外的是,特定细胞类型的基因表达模式高度依赖于局部组织背景。
这些实验结果表明,seqFISH+能够很好地绘制组织中的转录组,克服了光学拥挤,展现了seqFISH+在组织中生成空间单细胞图谱的强大应用潜力。与现有方法相比,seqFISH+在每个细胞中检出的mRNAs数目和RNA条形码总数扩大了10倍以上,它让人们得以使用普通共聚焦显微镜进行超分辨成像,并有能力推广至染色体和蛋白质成像。
“它的高通量基因组覆盖率和空间分辨率是阐明细胞间信号相互作用,理解发育过程和细胞命运决定的重要武器,最后,还有助于我们发现疾病样本中特定细胞类型的靶点,为精确的空间基因组学和单细胞诊断打下坚实基础,”文章作者总结道。