在我们日常生活中,拍照是记录生活的常用手段,我们的日常拍照设备也从老式的“傻瓜”相机逐渐发展成为了轻巧实用的数码相机。相片所反映出来的是我们所生活的客观世界的一草一木、一人一景,我们可以看见轮廓和外在,却看不到内部的结构,为了更好地探索世界,也为了更好地了解生物体的奥秘,科学家在漫长的历史中发明出了很多种用于“窥探”这个世界内部结构的成像方法,比如有电子显微镜(Electron Microscope,EM)、光学显微镜(Optical Microscope,OM)、核磁共振(Magnetic ResonanceImaging,MRI)、计算机断层扫描(Computed Tomography,CT)等医学常用手段;还有荧光成像(Fluorescence Imaging)、原子力显微镜(Atomic Force Microscope,AFM)、扫描隧道显微镜(Scanning Tunnel Microscope,STM)成像等新方法。
这些技术虽然已经极大地促进了医学和科学的发展,但是在应用方面还有很多的不足,常用的医学成像手段分辨率较低,在医学检测的应用上有很大的限制;而分辨率较高的荧光成像技术需要使用荧光物质标记,从而会改变目标分析物所在的理化环境,在临床应用上有潜在的误差;发展迅速的原子力显微镜和扫描隧道显微镜虽然已经解决了分辨率低的问题,但是在应用上仍存在无法对被测物进行定性分析的弊端。
本文想给大家介绍的质谱成像(Mass Spectrometry Imaging,MSI)技术,不需要特异性标记,样品前处理步骤简单,空间分辨率高,同时还能发挥质谱的效能,在进行目标物成像的同时提供目标物的结构信息。得益于质谱技术的发展,质谱成像技术已经可以在较宽的质荷比范围内对小分子及大分子进行成像分析,并且受到了各领域科学家的广泛关注,也在近20年内取得了极大的发展。那么,究竟什么是质谱成像呢?
当我们看到一张数码相片的时候,我们会知道这张相片是由红绿蓝三种颜色通道叠加而成的,色彩所拼接出的图像就是我们所生存的客观世界的二维表象。对于质谱成像而言,每一张照片是由成千上万个颜色通道叠加在一起的,每一种颜色通道所代表的是研究人员想要了解的一种目标分子,颜色的深浅代表着该物质的丰度,当这些通道互相覆盖叠加在一起,就会形成颜色各异、深浅不一的图像,进而反映出被研究分子的空间分布。
截止到目前为止研究人员为质谱成像设计了几十种方案,最常用、发展较好的有三种[1, 2],它们是次级离子质谱成像技术(Secondary Ion Mass Spectrometer-Mass Spectrometry Imaging,SIMS-MSI)、基质辅助激光解析质谱成像技术(Matrix-Assisted Laser Desorption Ionization-Mass Spectrometry Imaging,MALDI-MSI)和电喷雾解析电离成像技术(Desorption Electrospray Ionization-Mass Spectrometry Imaging,DESI-MSI),我们将对这三种方法进行进一步的了解。
这三种方法的原理虽各有不同,但都是在利用质谱进行质荷比(m/z)测定的基础上,结合特定的成像软件,对待测物不同部位的目标分子进行检测、鉴定、成像、叠加,进而得到反映待测物含量的空间分布情况的图像。
在质谱检测中,想要通过质量分析器得到样品中各类分子的m/z的信息,就要使被研究物质的表面解离和离子化。不同的质谱方法有着不同的手段,其中,SIMS-MSI主要利用初级离子束轰击待测品表面,再将从表面溅射出来的次级离子引入质量分析器进行质量分离和测定;而MALDI-MSI主要利用基质吸收激光能量,进而将能量传递给样品分子并使之电离;DESI-MSI则利用被雾化的带电液滴撞击样品表面,样品在被高速液滴撞击后发生溅射,进而进行质谱分析,得到质谱图。
软件对所得到的质谱峰进行识别,选定目标分子,根据该物质在样品不同位置处的相对出峰强度确定其含量分布,通过颜色深浅进行反映,便能得到该物质的成像图。对不同颜色图像进行叠加,便得到完整的成像图。
我们将在正文进一步对三种方法的发展及应用进行详细的介绍。
次级离子质谱法(SIMS)成像是质谱成像方法中具有高分辨率的,应用最早的方法之一,它第一次跟世人见面是在20世纪80年代[3]。与其他MSI技术相比,SIMS的主要优势是能够将分析区域限制在~100 nm,使用铯或氧一次离子束,可以使光斑聚焦至~50 nm,通过离子束的优化,能实现迄今为止MSI方法的最高空间分辨率。次级离子质谱成像主要利用一束初级离子(如:Ga+、Bi+、Bi3+、Bi52+、C60+、Au+、Au+n、Ar+n、Cs+n、SF5+等)喷射待测样品表面,从而使表面的原子带电,进而通过磁场、电场进行质谱分析和成像,来精确探测待测物的表面信息。但是由于初级离子束的能量通常较高,容易使共价键发生断裂,在测试时常得到大量的碎片离子。因而,SIMS通常适用于分析无机材料以及在表面通过化学键结合的材料,可用于元素分析、无机分析以及小分子物质(< 1000 Da)的检测[4]。
为了适应越来越广泛的大分子检测需求,SIMS-MSI在近来年注重于软电离源的开发,通过选用新型的聚集离子束大大地减少了样品的表面损伤和碎片化[5]。
2005年,Winograd教授[6]在Anal. Chem.杂志上发表了一篇名为The Magic of Cluster SIMS的文章,阐述了用C60+作为聚集离子束进行检测的优点。当许多原子参与表面的碰撞时,每个原子携带的能量成比例地减少,并且能够将入射能量沉积在更接近表面的位置,减少对样品表面的损伤,同时有效地刺激原子对能量的解吸[7]。Au+n[8, 9]等聚离子团簇,Ar+n[10, 11]等气体团簇的研究也有了一定的进展。2013年,英国曼彻斯特大学的Vickerman教授等[12]提出,来自有机和生物分子的低二次离子产率是未来利用TOF-SIMS作为表面材料分析技术的主要限制因素,因此,该团队使用超音速射流法开发了原型水簇离子束,在高离子剂量的条件下测量了四种标准化合物:精氨酸、氟哌啶醇、DPPC和血管紧张素的二次离子产率,与氩束相比,四种化合物都有更高的二次粒子产率,这些结果也鼓励了科学家去发展更高能量和更大尺寸的水簇束来进一步增强二次离子产率,使SIMS-MSI有更广泛的应用。
近年来,利用串联质谱与SIMS-MSI结合,对检测到的分子进行了更加可靠的结构测定[13, 14]。2016年,Fisher等[15]报告了一种使用新的TOF-SIMS与MS/MS并行成像的仪器。该技术结合了具有高分辨率的待测分子识别方法,能够在TOF-SIMS成像同时对复杂的化学成分进行筛选,并通过MS/MS定向鉴定相应成分。与传统的串联质谱方法相比,这种方法最大限度地利用了多组分样品产生的所有离子,目前研究人员已将这种方法应用于纳米尺度的分子表面分析和分子鉴定。
SIMS相对于其他MSI技术的另一个关键优势是可以利用初级离子束逐渐去除材料表面,不断对新暴露的表面进行检测成像,通过不断循环该过程可以实现对于分析物的3D分析(即深度剖析),其深度分辨率可以低至几纳米[16]。Goor等[17]在最近的一篇文章中,使用3D-SIMS-MSI对超分子组装体内的掺入材料的空间分布进行了成像。作为热塑性弹性体的脲基嘧啶酮(UPy)基体与反应性UPy-四嗪(UPy-Tz)添加剂混合后,UPy-Tz将在UPy基体上形成一个功能化表面。为了探究使UPy表面功能化的最适UPy-Tz浓度,研究人员在100 μm × 100 μm的区域内对UPy-Tz中的特定氟信号进行3D-SIMS成像,直接观察表面和基体内部的反应性的差异,确认最佳的功能化条件。如图1所示,利用3D-SIMS-MSI技术,便可以得到不同浓度的UPy-Tz添加剂在UPy基体内的分布的成像结果,进而更加直观地指导下一步实验。

图1 不同浓度的UPy-Tz在UPy基体内的3D-SIMS成像[17]
截至目前,SIMS-IMS已经广泛应由于生命科学研究的各个领域,对于微生物生态学[18, 19]、基因芯片检测[20, 21]、细胞生物学[22]、代谢组学研究[23, 24]、药物发现及分布研究[25]有着重大的意义。
虽然SIMS-MSI已经得到了较快的发展,但是它对于生物大分子的检测效果仍不尽如人意。为了克服这一挑战,科学家研究并建立了MALDI-MSI。该方法在1994年第42届美国质谱学会(ASMS)会议期间被首次提出,后来在1997年被成功应用[26],现已被广泛应用于植物、昆虫及哺乳动物组织的成像分析[26-29]。
1997年,Caprioli等[26]首次将MALDI-MSI应用于生物组织,并且成功实现了对大鼠脑垂体中激素肽的成像,证实了通过MALDI-MSI可以直接获得未经前处理和特异性标记的生物组织的成像谱图。此后,该项技术在生命科学研究领域得到越来越广泛的关注。
由于蛋白质在建立缺陷细胞间的信号联通途径的过程中承担着重要的作用,因而,对病理部位的蛋白质进行空间定位和分辨,进而监测组织的病变情况对于临床医学和生命科学具有重要的意义,而MALDI-MSI的发展使得蛋白质的空间成像变得可行,可协助科学家选择合适的疾病标志物,有利于疾病的早期发现和监测。但由于MALDI产生的离子通常具有低电荷状态(≤ 3),大大降低了它们的气相碎裂效率,因而在成像检测过程中能覆盖的蛋白质序列很有限[30, 31]。为了克服这些挑战,目前研究人员通常在成像数据采集之后进行独立的、互补的蛋白质组学实验。先从组织中提取蛋白质,随后通过电喷雾电离(ESI)分析样品,通过更传统的蛋白质组学工作流程进行蛋白质鉴定和精确的质量匹配,并将得到的鉴定结果与成像数据相关联,从而实现更准确、完善的蛋白质组学分析[32-34]。MALDI-MSI还可以用来监测组织中异生素及其代谢物的分布,成为药物分布及生命科学研究中的宝贵工具,图2所表示的便是MALDI-MSI在进行成像时的具体流程及原理[35, 36]。
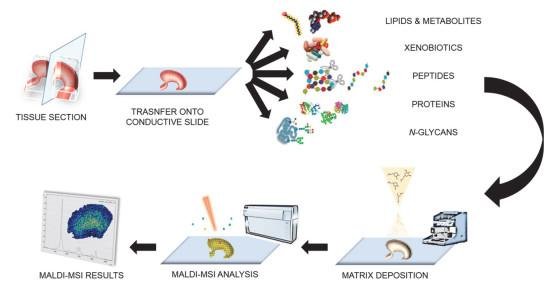
图2 MALDI-MSI成像工作原理图[36]
为了进一步优化MALDI-MSI的成像效果,科学家在二维拍照技术的基础上不断探索更高维度的成像手段。美国爱荷华州立大学的研究人员使用MALDI-MSI对新受精的斑马鱼胚胎单细胞中磷脂类——磷脂酰胆碱(PC)、磷脂酰乙醇胺(PE)以及磷脂酰肌醇(PI)进行了可视化的三维空间分布检测。这是MALDI-MSI首次应用于单个细胞的三维化学成像。相关研究成果已经发表在Scientific Reports上[37]。
在具体的实验中,典型的MALDI-MSI分析常在安装到导电载玻片上的组织切片上进行[38],蛋白质、脂质、异生素和代谢物的成像,需要在新鲜冷冻的组织切片上进行[39, 40]。目前,该技术在保持极高的灵敏度的基础上,实现了高达1000像素/秒的成像速率,5–10 µm的高空间分辨率,使得质谱成像可真正应用于临床医学中。但我们不可忽视的是,MALDI基质在吸收紫外线(UV)或红外线(IR)辐射激光的能量以解吸和电离基质中的分析物方面起着关键作用,正确选择和应用MALDI基质对MALDI-MSI至关重要,直接影响分析物的电离效率和组织成像的空间分辨率[41]。以往常用的MALDI质谱基质都为有机小分子,如2, 5-二羟基苯甲酸(DHB),α-氰基-4-羟基肉桂酸(CHCA),芥子酸(SA),2, 5-二羟基苯乙酮(DHAP),9-氨基吖啶(9-AA)和2-巯基苯并噻唑(2-MBT)。
其中,SA和DHAP通常用于检测高分子量的生物分子,例如蛋白质、糖蛋白和寡糖;CHCA和2-MBT通常适用于检测中等分子量的分析物,例如肽和脂质;DHB和9-AA已被证明是分析低分子量的化合物(如葡萄糖)的有效基质,如:氨基酸、脂肪酸和核苷酸。传统的有机基质虽然在成像方面也有一定的潜力,但其通常在低质量范围内有较高的干扰信号,在负离子检测模式中电离效率较低,目前研究人员已经开发出了多种新型基质以克服传统基质的弊端。新型的有机基质如中国科学院聂宗秀团队开发所开发的N-(1-萘基)乙二胺二盐酸盐[42],可用于脂质的检测的4-苯基-α-氰基肉桂酸酰胺[43]及二噻醇[44]等,食品添加剂中常用的植物提取物——姜黄素在2018年被证明是一种可用作MALDI质谱成像的优良基质,成功应用于药物、皮肤和肺组织中的脂质以及指纹中的多种化合物的分析及成像[45]。正如图3所示,郭寅龙团队[46]通过进行待测物衍生化,在正离子模式下实现了甲状腺癌组织中磷脂和被衍生化的游离脂肪酸的同时检测及成像分析,为更好地了解癌组织中的新陈代谢过程提供了新的方法。
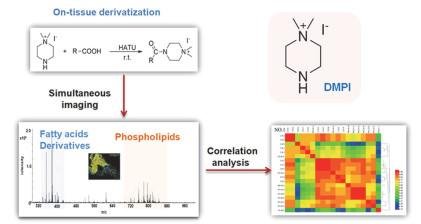
图3 同时检测甲状腺癌组织中磷脂和被衍生化的游离脂肪酸的示意图[46]
解吸电喷雾电离质谱成像(DESI-MSI)是于21世纪被开发的[47],最年轻的非破坏性质谱成像技术,可以对组织切片中的小分子进行原位分析,是一种快速的、大气压环境下的质谱成像技术。该方法几乎不需要对样品进行任何的前处理,因此可以反映出被检测物在生物组织中的原始分布及含量。2006年,普渡大学的Cooks教授团队[48]在没有任何前处理的情况下,在大气压力下对大鼠脑组织切片进行了检测,并通过DESI-MSI技术绘制脂质的分布图像,空间分辨率小于500 μm,如图4所示,通过解吸电喷雾电离质谱检测大鼠脑组织得到的质谱图,选定目标分子(m/z 327)经过成像软件分析,便可以得到该分子在待测组织中的分布。
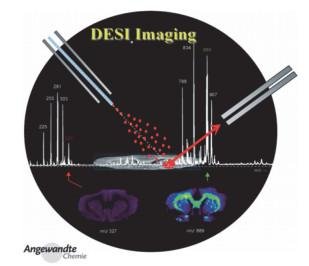
图4 DESI-MSI成像工作原理图[48]
2008年,该团队将解吸电喷雾电离(DESI)与线性离子阱质谱仪耦合,用于直接从脑、肺、肾和睾丸的组织切片记录药物的空间分布。DESI检测的结果与液相色谱-质谱/质谱联用(LC-MS/MS)检测结果相一致,有利于使用DESI成像直接从生物组织中检测药物和代谢物[49]。2010年,Cooks教授将其应用于人体晶状体中脂质分布的测定[50],并在2016年实现了人脑组织切片中肿瘤组织的成像分析[51]。
由于该项技术可以适用于大气环境下,对被检测组织无需前处理,在近十年内也在植物研究的领域得到了广泛的应用,Ifa团队将其应用于植物组织在病原体入侵时产生的代谢反应的快速成像中[52],Pradeep团队[53]使用该技术实现了对植物的种子、果皮、茎中的色酮生物碱的检测。
目前,DESI仍存在一些不足,样品表面分子的溶解和电离会受到电喷雾溶剂的组成不同的影响,同时,受其喷雾的空间分辨率的限制,DESI成像的分辨率一般约为200 μm,与SIMS和MALDI-MS相比,分辨率仍较低。
在这数十年间,质谱成像吸引了众多研究人员的目光,也得到了快速的发展。但是质谱成像技术到目前为止仍存在很多问题,比如空间分辨率受限,检测灵敏度不够高,对操作人员的专业素养要求较高不利于大规模的商业应用,对于神经递质等重要的生命小分子的检测效果还有待提高,对于植物体表面及内部组织的次生代谢物、活性成分等的检测还有很大的发展前景。我们能看到过去20年质谱成像技术的突破性进展和面临的挑战,同时我们也相信未来的研究会赋予质谱成像技术更多的可能性。
参考文献
[1] Dihazi H. ; Bohrer R. ; Jahn O. ; Lenz C. ; Majcherczyk A. ; Schmidt B. ; Urlaub H. ; Valerius O. ; Asif A. R. Expert Rev. Proteomics 2013, 10 (1), 17.
[2] Vachet R. W. Nat. Nanotechnol. 2015, 10, 103. DOI:10.1038/nnano.2015.4
[3] Briggs D. Surf. Interface Anal. 1983, 5, 113. DOI:10.1002/sia.740050307
[4] Lechene C. ; Hillion F. ; McMahon G. ; Benson D. ; Kleinfeld A. M. ; Kampf J. P. ; Distel D. ; Luyten Y. ; Bonventre J. ; Hentschel D. ; et al J. Biol 2006, 5, 20. DOI:10.1186/jbiol42
[5] Winograd N. Anal. Chem. 2015, 87 (1), 328.
[6] Winograd N. Anal. Chem. 2005, 77 (7), 142A.
[7] Mahoney C. M. ; Roberson S. V. ; Gillen G. Anal. Chem. 2004, 76 (11), 3199. DOI:10.1021/ac035532n
[8] Touboul D. ; Kollmer F. ; Niehuis E. J. Am. Soc. Mass Spectrom 2005, 16 (10), 1608. DOI:10.1016/j.jasms.2005.06.005
[9] Li Z. ; Verkhoturov S. V. ; Schweikert E. A. Anal. Chem. 2006, 78 (21), 7410. DOI:10.1021/ac0603779
[10] Rabbani S. ; Barber A. M. ; Fletcher J. S. ; Lockyer N. P. ; Vicerman J. C. Anal. Chem. 2011, 83 (10), 3793. DOI:10.1021/ac200288v
[11] Ninomiya S. ; Nakata Y. ; Honda Y. ; Ichiki K. ; Seki T. ; Aoki T. ; Matsuo J. Appl. Surf. Sci. 2008, 255 (4), 1588. DOI:10.1016/j.apsusc.2008.05.004
[12] Rabbani S. S. ; Barber A. ; Fletcher J. S. ; Lockyer N. P. ; Vickerman J. C. Anal. Chem. 2013, 85 (12), 5654. DOI:10.1021/ac4013732
[13] Fletcher J. S. ; Rabbani S. ; Henderson A. ; Blenkinsopp P. ; Thompson S. P. ; Lockyer N. P. ; Vickerman J. C. Anal. Chem. 2008, 80 (23), 9058. DOI:10.1021/ac8015278
[14] Carado A. ; Passarelli M. K. ; Kozole J. Anal. Chem. 2008, 80 (21), 7921. DOI:10.1021/ac801712s
[15] Fisher G. L. ; Bruinen A. L. ; Potocnik N. O. ; Hammond J. S. ; Bryan S. R. ; Larson P. E. ; Heeren R. M. A. Anal. Chem. 2016, 88 (12), 6433. DOI:10.1021/acs.analchem.6b01022
[16] Wucher A. ; Cheng J. ; Zheng L. ; Winograd N. Anal. Bioanal. Chem. 2009, 393, 1835. DOI:10.1007/s00216-008-2596-5
[17] Goor O. J. G. M. ; Keizer H. M. ; Bruinen A. L. ; Schmitz M. G. J. ; Versteegen R. M. ; Janssen H. M. ; Heeren R. M. A. ; Janssen H. M. ; Heeren R. M. A. ; Danker P. Y. W. Adv. Mater. 2017, 29, 1604652. DOI:10.1002/adma.201604652
[18] Dekas A. E. ; Poretsky R. S. ; Orphan V. J. Science 2009, 326 (5951), 422. DOI:10.1126/science.1178223
[19] Lechene C. P. ; Luyten Y. ; McMahon G. ; Distel D. L. Science 2007, 317 (5844), 1563. DOI:10.1126/science.1145557
[20] Mayali X. ; Weber P. K. FEMS Microbiology Ecology 2018, 94 (5), fiy047.
[21] Rao A. N. ; Vandencasteele N. ; Gamble L. J. ; Grainger D. W. Anal. Chem. 2012, 84 (24), 10628. DOI:10.1021/ac3019334
[22] Steinhauser M'. L. ; Bailey A. P. ; Senyo S. E. ; Guillermier C. ; Perlstein T. S. ; Gould A. P. ; Lee R. T. ; Lechene C. P. Nature 2012, 481 (7382), 516. DOI:10.1038/nature10734
[23] Ostrowski S. G. ; Van Bell C. T. ; Winograd N. ; Ewing A. G. Science 2004, 305 (5680), 71. DOI:10.1126/science.1099791
[24] Lozano M. M. ; Liu Z. ; Sunnick E. ; Janshoff A. ; Kumar K. ; Boxer S. G. J. Am. Chem. Soc. 2013, 135 (15), 5620. DOI:10.1021/ja310831m
[25] Passarelli M. K. ; Newman C. F. ; Marshall P. S. ; West A. ; Gilmore I. S. ; Bunch J. ; Alexander M. R. ; Dollery G. T. Anal. Chem. 2013, 87 (13), 6696.
[26] Caprioli R. M. ; Farmer T. B. ; Gile J. Anal. Chem. 1997, 69 (23), 4751. DOI:10.1021/ac970888i
[27] Kompauer M. ; Heiles S. ; Spengler B. Nat. Method 2017, 14 (1), 90. DOI:10.1038/nmeth.4071
[28] Qin L. ; Zhang Y. W. ; Liu Y. Q. ; He H. H. ; Han M. ; Li Y. Y. ; Zeng M. M. ; Wang X. D. Phytochem. Anal. 2018, 29 (4), 251.
[29] Pratavieira M. ; Silva Menegasso A. R. ; Esteves F. G. ; Sato K. U. ; Malaspina O. ; Palma M. S. Phytochem. Anal. 2018, 17 (7), 2358.
[30] Boersema P. J. ; Taouatas N. ; Altelaar A. F. M. ; Gouw J. W. ; Ross P. L. ; Pappin D. J. ; Heck A. J. R. ; Mohammed S. Mol. Cell Proteom. 2009, 8 (4), 650. DOI:10.1074/mcp.M800249-MCP200
[31] Mekecha T. T. ; Amunugama R. ; Mcluckey S. A. J. Am. Soc. Mass Spectrom. 2006, 17 (7), 923. DOI:10.1016/j.jasms.2006.01.004
[32] Alberts D. ; Pottier C. ; Smargiasso N. ; Baiwir D. ; Mazzucchelli G. ; Delvenne P. ; Kriegsmann M. ; Kazdal D. ; Warth A. ; Pauw E. D. ; Longuespee R. Proteom. Clin. Appl. 2018, 12 (1), 1700062. DOI:10.1002/prca.201700062
[33] Yajima Y. ; Hiratsuka T. ; Kakimoto Y. ; Ogawa S. ; Shima K. ; Yamazaki Y. ; Yoshikawa K. ; Tamaki K. ; Tsuruyama T. Sci. Rep. 2018, 8, 7493. DOI:10.1038/s41598-018-25817-7
[34] Spraggin J. M. ; Rizzo D. G. ; Moore J. L. ; Noto M. J. ; Skaar E. P. ; Caprioli R. M. Proteomics 2016, 16 (11-12), 1678. DOI:10.1002/pmic.201600003
[35] Aichler M. ; Walch A. Lab. Investig. 2015, 95 (4), 422. DOI:10.1038/labinvest.2014.156
[36] Smith A. ; Piga I. ; Galli M. ; Stella M. ; Denti V. ; Puppo M. D. ; Magni F. Int. J. Mol. Sci. 2017, 18 (12), 2588. DOI:10.3390/ijms18122588
[37] Duenas M. E. ; Essner J. J. ; Lee Y. J. Sci. Rep. 2017, 7, 14949. DOI:10.1038/s41598-017-15020-5
[38] Baker T. C. ; Han J. ; Borchers C. H. Curr. Opin. Biotechnol. 2017, 43, 62. DOI:10.1016/j.copbio.2016.09.003
[39] Goodwin R. J. A. ; Nilsson A. ; Borg D. ; Langrudge-Smith P. R. R. ; Harrison D. J. ; Mackay C. L. ; Iverson S. L. ; Andren P. E. J. Proteom. 2012, 75 (16), 4912. DOI:10.1016/j.jprot.2012.07.006
[40] Nilsson A. ; Goodwin R. J. A. ; Shariatgorji M. ; Vallianatou T. ; Webborn P. J. H. ; Andrem P. E. Anal. Chem. 2015, 87 (3), 1437. DOI:10.1021/ac504734s
[41] Thomas A. ; Chaurand P. Bioanalysis 2014, 6 (7), 967. DOI:10.4155/bio.14.63
[42] Wang J. ; Qiu S. ; Chen S. ; Xiong C. Q. ; Liu H. H. ; Wang J. Y. ; Zhang N. ; Hou J. ; He Q. ; Nie Z. X. Anal. Chem. 2015, 87 (1), 422.
[43] Fulop A. ; Porada M. B. ; Marsching C. ; Blott H. ; Meyer B. ; Tambe S. ; Sandhoff R. ; Junker H. D. ; Hopf C. Anal. Chem. 2013, 85 (19), 9156. DOI:10.1021/ac4018154
[44] Le C. H. ; Han J. ; Borchers C. H. Anal. Chem. 2012, 84 (19), 8391. DOI:10.1021/ac301901s
[45] Francese S. ; Bradshaw R. ; Flinders B. ; Mitchell C. ; Belay S. ; Cicero L. ; Clench M. R. Anal. Chem. 2013, 85 (10), 5240. DOI:10.1021/ac4007396
[46] Wang S. S. ; Wang Y. J. ; Zhang J. ; Sun T. Q. ; Guo Y. L. Anal. Chem. 2019, 91 (6), 4070. DOI:10.1021/acs.analchem.8b05680
[47] Takats Z. ; Wiseman J. M. ; Gologan B. ; Cooks R. G. Science 2004, 306 (5695), 471. DOI:10.1126/science.1104404
[48] Wiseman J. M. ; Ifa D. R. ; Song Q. Y. ; Cooks R. G. Angew. Chem. Int. Ed. 2006, 45 (43), 7188. DOI:10.1002/anie.200602449
[49] Wiseman J. M. ; Ifa D. R. ; Zhu Y. ; Kissinger C. B. ; Manicke N. E. ; Kissinger P. T. ; Cooks R. G. PNAS 2008, 105 (47), 18120. DOI:10.1073/pnas.0801066105
[50] Ellis S. R. ; Wu C. P. ; Deeley J. M. ; Zhu X. J. ; Truscott R. J. W. ; Panhuis M. ; Cooks R. G. ; Mitchell T. W. ; Blanksby S. J. J. Am. Soc. Mass Spectrom 2010, 21 (12), 2095. DOI:10.1016/j.jasms.2010.09.003
[51] Jarmusch A. K. ; Alfaro C. M. ; Pirro V. ; Hattab E. M. ; Cohen-Gadol A. A. ; Cooks R. G. PLOS ONE 2016, 11 (9), e0160380.
[52] Tata A. ; Perez C. J. ; Hamid T. S. ; Bayfield M. A. ; Ifa D. R. J. Am. Soc. Mass Spectrom. 2015, 26 (4), 641. DOI:10.1007/s13361-014-1039-0
[53] Kumara P. M. ; Srimany A. ; Arunan S. ; Ravikanth R. U. S. ; Pradeep T. PLOS ONE 2016, 11 (6), e0158099. DOI:10.1371/journal.pone.0158099
杨迎晨, 陈企发, 夏炎. 更深度的“照相”技术——质谱成像的发展与应用. 大学化学[J], 2020, 35(3): 47-53 doi:10.3866/PKU.DXHX201907028
Yang Yingchen. More in-Depth "Photography" Technology: The Development and Application of Mass Spectrometry Imaging. University Chemistry[J], 2020, 35(3): 47-53 doi:10.3866/PKU.DXHX201907028