SNaPshot®多重分析系统是一种基于引物单碱基延伸的方法,在单管反应中,能够对多个 SNP进行多重分析。利用此系统可筛选和验证 SNP、评估 DNA 甲基化、对 BAC 进行指纹识别、以及筛选样品的易感性等等。SNaPshot既拥有Sanger测序中ddNTP单碱基延伸就终止的特点,又具有片段分析通量较高的属性,如图1示例,若同时检测3个位点,首先通过PCR扩增含有SNP的目标区域,然后再通过四种不同荧光基团标记的ddNTP单碱基延伸,获得SNP的信息,最后通过毛细管电泳基因分析仪(图2中所有仪器上都可以完成)得到SNP检测结果。
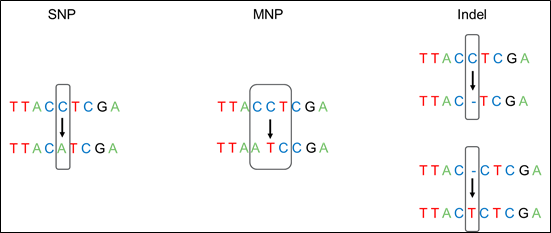
注:仪器型号从左往右,依次为3730系列、3500系列、3130系列、SeqStudio先澄清一个,这里所讲的“SNP”包括SNP,MNP以及Indel(如图3所示)。文章中后续提及的“SNP”,实际上是这个含义。
注:SNP(Single Nucleotide Polymorphism):单核苷酸多态性,一个碱基的变化。分为双等位的(Biallelic)和多等位的(Multiallelic);MNP(Multi-Nucleotide Polymorphism):相对于Reference,一般指多于一个的连续碱基变化(长度不变),有多个碱基的变化;Indel(Insertion/Deletion),插入/缺失,指相对于Reference,有单个或者是多个碱基的插入/缺失。在这里,不再展开讨论SNP的应用方向了,这个在网上、圈里、讲座等等有很多线索可帮您找到。在这儿,我们专注SNaPshot技术如何落地。
接下来,我们就一起来关注SNaPshot整个流程,包括如何设计引物、订购试剂、预实验、正式实验和最后分析数据(图4)。

- 引物设计:采用引物设计软件设计引物;评估引物之间是否存在引物二聚体,发卡结构等
- 订购试剂:DNA提取试剂、SNaPshot实验流程相关试剂
- 预实验:测试引物是否可用;并探索稳定的反应体系和反应条件
- 正式实验:把所有样本按照预实验的摸索,做完所有样本,并收集整理好数据
- GeneMapper数据分析:利用软件,call出所有SNP

可以在公共数据库:NCBI,Ensembl,UCSC等等中查找,或者是自己收集的参考序列。然后以SNP位点为中心,上下游至少包括 200bp的参考序列,以便引物设计软件查找合适的扩增引物。
这里必须先停下来,得让您先思考一下,想想您的目标是做单重还是做多重。若是没想好,可先来看下多重和单重的实验流程的对比。这里以3个SNP(SNP1,SNP2,SNP3)为示例,向您阐述两种流程的区别,另外,这里讲的“多重”是指SNaPshot实验流程的第一步PCR扩增含有靶标SNP位点的片段时,采用多重扩增方式扩增多个靶标片段,以达到节约试剂、提高效率和简化流程之目的(如图5)。
注:这里以3个SNP为例,左侧流程,核酸提取完后,3个SNP做同管同时扩增,也就是多重PCR扩增,接着产物纯化,再做SNaPshot反应,再纯化后,上机检测;右侧流程和左侧流程的区别在于第一步目标区域扩增这里,SNP位点单独分开扩增,也就是单重PCR扩增,再各自单独纯化,纯化的产物按照比例混匀之后再去做SNaPshot反应。如果SNaPshot反应,您打算只做单重的话,您必是壕无疑啦!如果您打算做多重,请接着往下看;如果您打算做单重,请先等待下,本文的下篇最后会有解释。
建议PCR产物长度范围至少在150bp-200bp,为了方便测序验证;
尽量使得靶标SNP位点位于片段的中间位置。这个主要是为了满足Sanger测序验证需要;
各个引物之间的GC含量、Tm值等等尽量接近,便于优化多重PCR扩增反应条件;
SNaPshot单碱基延伸引物设计:这个引物是比较好设计的,在SNP的左边,或者反向互补链(右边)选择17-25mer长度的序列即可,最后确认,自身是否有严重的发卡结构、引物二聚体(self-dimer),还要与其他单碱基延伸引物做分析,看是否有严重的引物二聚体(cross-dimer);
引物设计软件非常多,您可以根据自己的使用习惯来选择。如果您是一枚“小白”,这篇讲BatchPrimer3(You et al., 2008)在线引物设计软件的文献值得您一读。在合成引物之前,建议用软件评估一下引物之间是否存在严重的非特异性结合或者是引物二聚体。推荐Multiplex manager软件评估(Holleley and Geerts, 2009)。这里讲的修饰实际上就是在单碱基延伸引物的5’端加上尾巴序列,使不同位点的延伸产物有大小的区别,最终可以通过毛细管电泳区分开,达到准确检测。
- 适合添加到引物5’端的尾巴序列基本上都采用重复序列,有 poly (dT)、poly (dA)、poly (dC)和poly (dGACT);
- 强烈建议:当引物长度小于36mer时,需做预实验,识别其实际的电泳大小位置,因为引物在毛细管电泳时,迁移受到引物长度、碱基组成、标记的荧光染料等因素影响。而引物越短,越易受到其碱基组成的影响,越有可能偏离原来预设的大小位置;
- 引物之间的长度(是指包括尾巴序列在内)差异建议最好在8到10个碱基,至少4到6个碱基;
- 引物的退火温度(Tm值,不包括尾巴序列)尽可能地一致,但都不能低于50℃;
- 要检查引物之间是否存在错配,避免出现非特异性结果;

欢迎您选择赛默飞引物合成,详情咨询:400-820-8982。注意:以上试剂多有不同包装规格,详寻当地销售或者在www.thermofisher.com官网上查询。

良好的预实验设计就已成功了一半。小编认为这句话就是为预实验量身定做的。
核酸样本推荐OD260/OD280的比值在1.8到2.0之间
该步骤的验证非常关键,主要分三部分完成。第一部分,得到每个位点的SNaPshot结果;第二部分,用Sanger测序验证每一个位点的结果是否和SNaPshot的结果一致;第三部分,做多重测试,验证多重PCR以及多重SNaPshot反应的最后结果与单个位点结果一致。
先把SNP位点的正反向引物取部分混匀,在引物的混合物中,每个引物的浓度为0.8 µM。例如,正向引物为10 µM,反向引物为10 µM,可按照如下比例混匀:
PCR反应体系配制:

PCR反应条件:
注意:[1] 如果引物的Tm值不在60-65℃,可能需要优化该处PCR退火温度PCR结束之后,从PCR仪中取出,离心;
开盖加入4 µL ExoSAP-IT™ Reagent,封盖,震荡混匀,离心;
反应条件
反应体系配制:
注:[1] 预先准备单碱基延伸引物混合物,每个引物在混合物中终浓度是0.2 μM反应条件:
反应体系配制:
反应条件:
体系配制:

Sanger测序流程可以参见文档 Publication Number: MAN0015798 (在www.thermofisher.com 网上搜索 “MAN0015798” , 即可找到该文档)。可以从 “第一部分 得到每个SNP位点的SNaPshot结果” 的PCR产物纯化后开始(图6)。得到测序结果后,与上面SNaPshot结果对比验证。请参见文档Publication Number:MAN0015798的第9-14页。

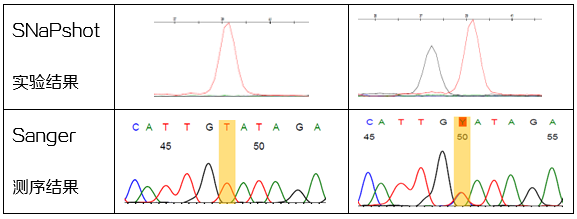
如下图7,该SNP位点是C/T,上面为SNaPshot结果,下面为对应PCR产物的测序结果,左侧为纯合的TT,右侧为杂合的CT(SNaPshot反应中,C用黑色表示,Sanger测序反应中,C用蓝色表示)。如果SNaPshot结果与Sanger测序结果一致,那么就可以开始多重实验测试了。多重实验的预实验怎么做、正式实验如何开展以及数据分析且待下回详解。非常感谢您的关注。
- You FM, Huo N, Gu YQ, Luo MC, Ma Y, Hane D, LazoGR, Dvorak J, Anderson OD. BatchPrimer3: a high throughput web application for PCR and sequencing primer design. BMC Bioinformatics. 2008,9:253, doi:10.1186/1471-2105-9-253
- Holleley CE and Geerts PG. Multiplex Manager1.0: a cross platform computer program that plans and optimizes multiplex PCR, BioTechniques. 2009, 46(7):511-517
- Protocol: ABIPRISM® SNaPshot™ Multiplex Kit
- User Bulletin: Generating high‑quality data using the BigDye™ Terminator v3.1 Cycle Sequencing Kit Using ExoSAP-IT™ Reagent for PCR purification, Publication Number MAN0015798, Revision A.
- User Guide: DNA Fragment Analysis by Capillary Electrophoresis, Publication Number 4474504, Revision B.
- User Guide: Platinum™ Multiplex PCR Master Mix, Publication Number 4463722, Revision B.