提取序列
当转录组和蛋白组数据经过一系列生信分析以后,筛选得到一些关注的基因和蛋白,想要对这些基因和蛋白进行深入挖掘,首先就是要获取这些序列信息,在没有生信基础的情况下,通常我们会根据目标ID信息去基因组文件中手动查找。目标基因或蛋白的数量较少的情况下,手动查找勉强可行。如果目标基因或蛋白的数量成百上千,手动查找的任务就很艰巨了,除了会耗费大量的时间和精力,同时也会因为数据量庞大而提高出错的概率。因此,迈维代谢云平台开发了《提取序列》小工具,可帮您快速批量提取基因和蛋白序列。
视频解说教程
迈维云链接:https://cloud.metware.cn
1
提取原理
物种基因组一般会对应一个fa文件和一个gff文件,fa文件里面就是对应的基因序列,gff文件是基因的结构信息,主要描述了基因组上各种特征的区间信息,包括染色体,基因,转录本等,基因的结构是基因组后续功能研究的基石。结合基因组的结构信息和fa序列信息,我们就可以提取目标基因的CDS序列,mRNA序列和蛋白序列。
fa文件示例
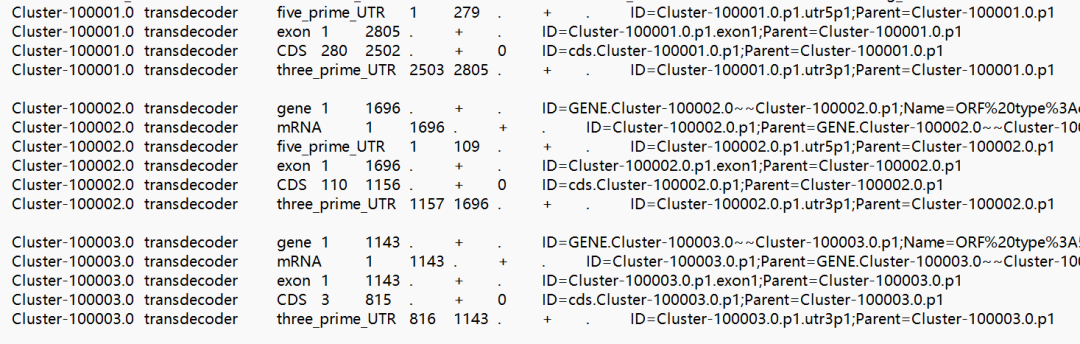
gff文件示例
2
适用物种
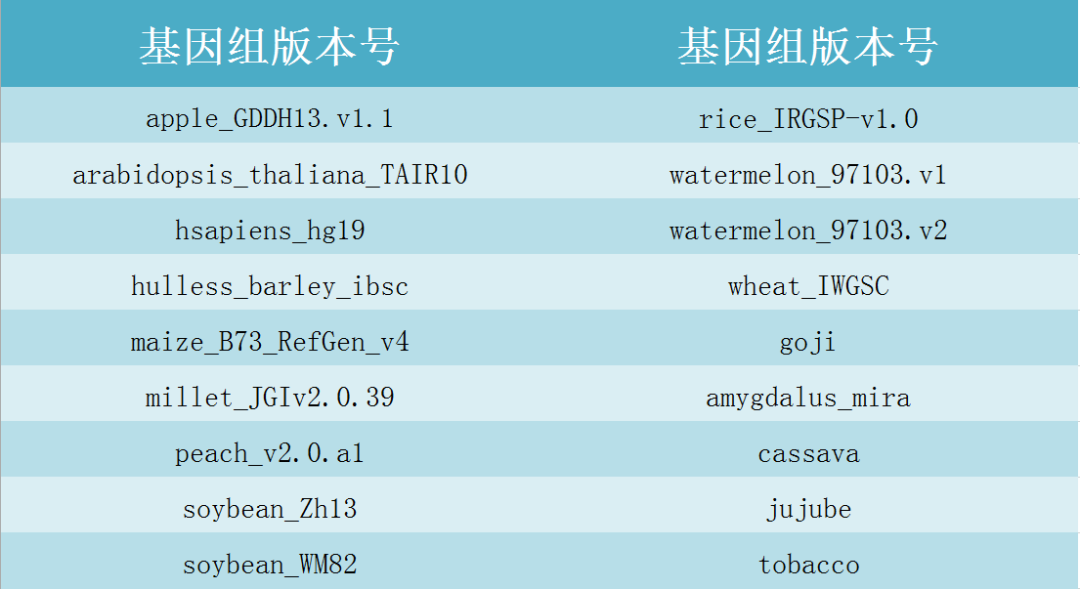
小工具目前已收纳18个基因组信息,包括每个基因组的fa序列和gff信息。用户无需上传基因组信息,选择基因组对应版本号即可。云平台已收录基因组版本号信息如下表:
3
三.输入文件
想要快速获取目标序列,您只需要准备一个文件,把您需要提取序列的基因ID整理到txt文档就可以了,部分基因ID如下表所示。如果ID名称需要100%匹配上就选精确匹配,例如LOC_Os01g01010.1就只能匹配上LOC_Os01g01010.1的ID序列,如果不需要100%匹配上就可以选模糊匹配,例如LOC_Os01g01010可以匹配上LOC_Os01g01010.1,LOC_Os01g01010.2,LOC_Os01g01010.3等所有包含LOC_Os01g01010的ID。
4
结果展示
任务运行结束后,会得到5个文件,以水稻rice_IRGSP-v1.0基因为例,输出的结果文件分别是2个txt文档:gene和readme;3个fa文件:rice_IRGSP-v1.0_cds、rice_IRGSP-v1.0_mRNA、rice_IRGSP-v1.0_prot。gene是输入的基因ID信息,readme是结果文件的说明文档,rice_IRGSP-v1.0_cds是CDS序列,rice_IRGSP-v1.0_mRNA是转录本序列,rice_IRGSP-v1.0_prot是蛋白序列。
往期精彩:
●视频实操SCI作图课(11):如何一眼看透你的数据分布——直方图
●视频实操SCI作图课(10):多组学分析必会——Kegg富集分析小工具
●视频实操SCI作图课(9):如何利用upsetR快速提取多组交集信息
●视频实操SCI作图课(8):一秒看懂数据集逻辑关系—高级韦恩图
●视频实操SCI作图课(7):多组学数据挖掘难?相关性聚类热图说so easy!
●视频实操SCI作图课(6):学会这个方法,让你的聚类热图卷起来
●视频实操SCI作图课(5):如何合并代谢物数据?合并表格2min搞定!
●视频实操SCI作图课(4):高级火山图实操,让你的文章火起来!
PLS-DA分析,组间差异的挖掘神器

微信:metware888
电话:027-62433042
邮箱:support@metware.cn
网址:www.metware.cn